Actualmente existe una crisis que nos tiene a todos los científicos un poco angustiados. La resistencia de patógenos microbianos está a la orden del día y se ha convertido en una prioridad mundial encontrar nuevos fármacos que funcionen. Esta desesperación ha llevado a un investigador español a mirar hacia una fuente inesperada: los organismos extintos. En este artículo, queremos acercaros a un avance muy original que une la paleogenómica con bioinformática e inteligencia artificial (IA): la desextinción molecular, un enfoque pionero liderado por el laboratorio del Dr. César de la Fuente, en la Universidad de Pensilvania.
¿Qué es la desextinción molecular?
La idea principal de la desextinción molecular es traer de vuelta a la vida el potencial terapéutico de especies extintas a partir de sus secuencias genómicas. Gracias a reconstrucción computacional con mapeadores y modelos de IA, el grupo de de la Fuente analiza proteomas extintos para identificar péptidos con propiedades antimicrobianas desconocidas en la actualidad, que posteriormente se sintetizarían artificialmente.
APEX: la IA al servicio de la lucha
Una de las herramientas más importante del equipo es el uso de APEX (Antibiotic Peptide de-Extinction), un sistema de deep learning entrenado para predecir estos péptidos antimicrobianos a partir de proteínas ancestrales. Su rápido análisis masivo de secuencias le hace capaz de seleccionar aquellas con características estructurales y fisicoquímicas asociadas a la actividad antimicrobiana en tiempo récord.
De esta forma, renacieron la mamutusina-2, derivada del mamut lanudo, y la megalocerina-1, del alce gigante Megaloceros giganteus. Ambos péptidos mostraron una gran capacidad bactericida frente a patógenos multirresistentes.
¿Por qué buscar antibióticos en especies extintas?
La biodiversidad extinta presenta una biblioteca molecular inexplorada. Muchas de estas especies desarrollaron defensas químicas únicas para protegerse de infecciones en entornos que hoy están desaparecidos. Además, sus proteínas presentan una arquitectura poco común en los sistemas modernos, lo que podría traducirse en mecanismos de acción distintos a los antibióticos convencionales, evitando también así la dependencia de compuestos de origen natural que se usan actualmente, como los producidos por bacterias del suelo.
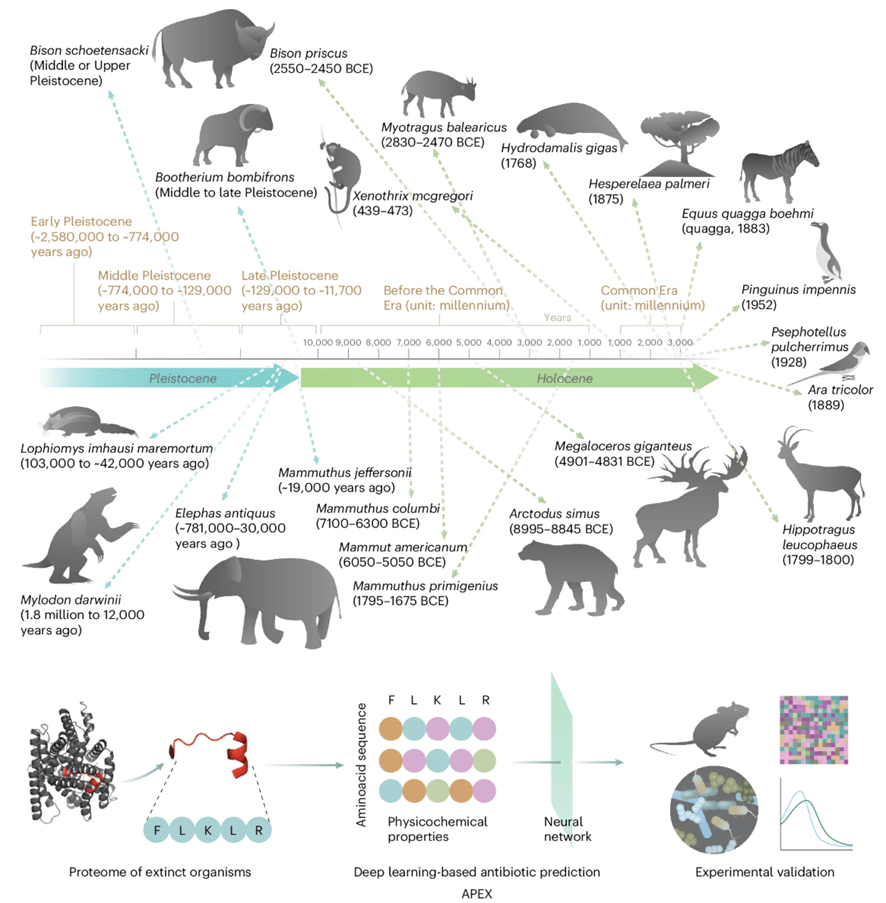
Validación experimental y eficacia in vivo
Los péptidos seleccionados por APEX no solo se sintetizan químicamente, sino que son evaluados en ensayos microbiológicos, modelos de infección y estudios de toxicidad, como se hace habitualmente con todos los nuevos fármacos. Algunos de ellos han demostrado una eficacia comparable a la polimixina B, un antibiótico de última línea, pero con menor toxicidad.
Además, estos péptidos muestran resistencia a las estrategias habituales de evasión bacteriana, lo que los convierte en candidatos prometedores frente a superbugs como Acinetobacter baumannii o Klebsiella pneumoniae.
Una nueva frontera en el descubrimiento de fármacos
El trabajo del grupo de César de la Fuente no solo representa un avance tecnológico, sino un cambio de paradigma en la biotecnología antimicrobiana. La combinación de la IA, junto con la síntesis peptídica y los datos genómicos antiguos abre un nuevo capítulo en la lucha contra las infecciones resistentes.
Si bien quedan muchas pruebas que realizar antes de que estos compuestos lleguen al uso clínico, la desextinción molecular sienta las bases para una estrategia de descubrimiento de antibióticos más diversa, evolutivamente informada y resistente a la obsolescencia.
CONCLUSIÓN
La desextinción molecular representa una prometedora estrategia biotecnológica para combatir la creciente amenaza de la resistencia microbiana. Al combinar paleogenómica, IA y la síntesis peptídica, esta línea de investigación abre nuevas posibilidades terapéuticas, aprovechando el legado genético de especies extintas para desarrollar antibióticos más eficaces y duraderos.
AUTORA
María Sánchez Carcelén
Responsable de la Unidad de Bioinformática
Hospital Nacional de Parapléjicos.
Toledo, España.
Referencia
Wan, F., Torres, M.D.T., Peng, J. et al. Deep-learning-enabled antibiotic discovery through molecular de-extinction. Nat. Biomed. Eng 8, 854–871 (2024). https://doi.org/10.1038/s41551-024-01201-x
Para más información, dudas o solicitud de presupuestos puedes contactarnos en:
msanchezcarcelen@externas.sescam.jccm.es
Síguenos en:
http://www.linkedin.com/in/servicios-de-apoyo-a-la-investigación-sais-hnp