La microbiota es el conjunto de microorganismos (bacterias, hongos, arqueas, virus y parásitos) que residen en nuestro cuerpo. La introducción de nuevas técnicas en el análisis de este ecosistema, como la citometría de flujo, supone un gran avance en el conocimiento de la composición de la microbiota y de su implicación en los estados de salud y enfermedad del ser humano.
MICROBIOTA INTESTINAL
El ecosistema microbiano más complejo de nuestro organismo es el asociado al aparato digestivo. Se estima que el intestino humano alberga un total de 1013 bacterias de 400 especies distintas y que llegan a alcanzar un peso de entre 300 y 600 gr. Su principal función es la de prevenir la colonización por otros microorganismos patógenos, ayudar a digerir los alimentos, producir vitaminas B y K que el organismo humano no es capaz de sintetizar y estimular al sistema inmune. Los cambios en la composición microbiana y, en consecuencia, los cambios en los metabolitos liberados, pueden tener gran impacto en el sistema inmunológico del huésped desencadenando una respuesta inflamatoria. Dichas alteraciones en la microbiota intestinal también se han relacionado con otras enfermedades del sistema nervioso central (autismo, ansiedad, depresión) y con enfermedades neurodegenerativas (Alzheimer, Parkinson, ELA, Enfermedad de Huntington), gracias a las evidencias científicas que han revelado la existencia de una comunicación bidireccional entre el microbioma intestinal y el sistema nervioso central, conocida como “eje microbiota-intestino-cerebro”. Este eje engloba el nervio vago, el sistema parasimpático, los metabolitos bacterianos, que pueden tener acciones como neurotransmisores, y el sistema endocrino asociado al tracto digestivo (Loh et al., 2024).
Tradicionalmente, para la determinación de la composición de la microbiota se han utilizado cultivos microbiológicos. Sin embargo, hoy en día se sabe que la mayor parte de los microrganismo de este ecosistema no se pueden cultivar con los medios habituales, siendo únicamente posible su detección tras la secuenciación de ADN como huella genética.
El conocimiento de este ecosistema se ha incrementado considerablemente tras la utilización de técnicas moleculares de secuenciación masiva, también conocidas como Next-generation Sequencing (NGS). La más utilizada es la amplificación del gen ADNr 16S, que está presente en la mayoría de las especies bacterianas y permite cuantificar la abundancia relativa de cada filo, familia o género. Sin embargo, este gen tiene diferente número de copias en cada género o especie por lo que este método presenta un gran inconveniente y es que tiende a sobreestimar la diversidad microbiana. Además, cabe destacar que esta técnica presenta un coste muy elevado y su estandarización resulta muy compleja.
El análisis de la microbiota por citometría de flujo no presenta esas desventajas, ya que nos permite caracterizar de manera individual las bacterias, analizar la heterogeneidad e incluso la abundancia relativa de cada especie debida a los cambios dinámicos que se producen en la composición y/o funcionalidad microbiana (disbiosis). Aunque no aporte directamente información filogenética, se pueden separar por citometría subpoblaciones bacterianas concretas para posteriores estudios moleculares que permitan determinar el papel de dichas bacterias.
HUELLA CITOMÉTRICA
Los citómetros de flujo de alta resolución permiten analizar la heterogeneidad y los cambios de la microbiota intestinal mediante la medida de la dispersión de la luz (Forwards Scatter) y el contenido de ADN. Para cuantificar el ADN se emplea el colorante 4′,6-diamidino-2′-fenilindol (DAPI), excitable por láser ultravioleta (355nm), que se une a regiones ricas en AT del ADN, permitiendo incluso resolver entre diferentes niveles cromosómicos.
El primer paso consistiría en la obtención de la muestra de la comunidad microbiana (Figura 1A). Esto puede ser, una muestra de heces o una muestra directa del microbioma intestinal. Estas muestras de microbioma (Figura 1B) son comunidades bacterianas diversas que constan de especies con diferentes antecedentes filogenéticos, características celulares y funciones. Para el análisis por citometría de flujo, el ADN de las bacterias se tiñe con DAPI (Figura 1C).
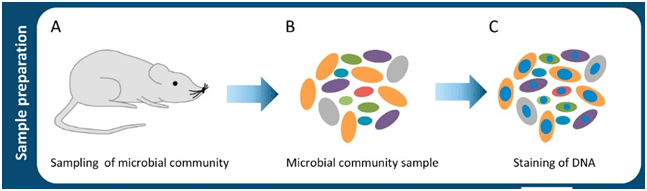
Figura 1. Esquema de los pasos a aseguir para la preparación de muestras en el análisis de microbiota por citometría de flujo. A) Recogida de muestras de la comunidad microbiana; B) Muestras microbiana; C) Tinción del DNA. Figura modificada de Koch et al. 2018.
Las muestras se adquieren por el citómetro de manera individualizada (Figura 2D). Los datos se visualizan en un gráfico biparamétrico, donde dependiendo de la señal de dispersión frontal (FSC) y la fluorescencia del ADN (DAPI), cada célula tendrá una posición específica en el gráfico y las células con características similares estarán más cerca unas de otras. De esta manera, el análisis por citometría representa la huella citométrica del microbioma (Figura 2E). Dicha huella representa la variedad de células con diferentes características ópticas, así como su abundancia en las respectivas subcomunidades (SC). Las SC se pueden identificar basándose en un análisis segregado y están agrupadas mediante gates o regiones (Figura 2F).
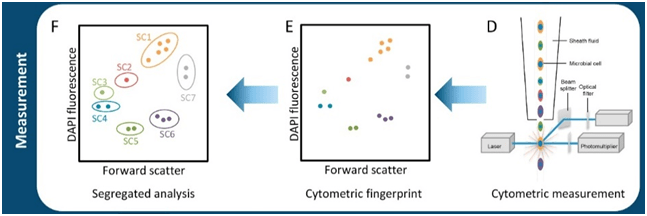
Figura 2. Flujo de trabajo en el análisis de microbiota por citometría de flujo. D) Adquición de muestras en el citómetro; E) Huella citométrica; F) Análisis segregado. Figura modificada de Koch et al. 2018
Recientemente, se han desarrollado una serie de herramientas bioinformáticas (flowCHIC, flowEMMi, FlowFP y flowCyBar) que permiten realizar análisis de rutina para revelar la disbiosis. Dichos cambios en la composición bacteriana se puede combinar con información funcional que categorice las muestras según su tratamiento específico, indicación clínica (sano o enfermo) u origen de la muestra (Figura 4G).
El CytBar o Cytometry Barcoding (código de barras citométrico), identifica las variaciones de la huella citométrica y la abundancia de las subcomunidades (SC) a lo largo del tiempo o debidas a factores experimentales (Figura 3H) mediante la aplicación de una plantilla que se aplica a todas las muestras (Figura 3).

Figura 3. Ejemplo de la elaboración de una plantlla de análisis mediante CytBar. Las poblaciones observadas en los dot plot (gráfico de puntos en citometría) se identifican con un gate o región tipo elipsoide. Se combinan los gates de todas las muestras (A y B en la imagen) para generar una plantilla (gate template) que finalmente se aplicará a todas las muestras, gracias a la cual obtendremos la abundancia de bacterias por gate y la indentificación de los cambios producidos en la composición y funcionalidad de las mismas.
Se pueden diferenciar los aumentos (tonos de rojo) y las disminuciones (tonos de azul) del número de bacterias en una SC. El análisis de correlación revela posibles funciones de las bacterias que conforman cada SC (Figura 4I). El análisis de co-ocurrencia revela interconexiones entre las SC individuales (Figura 4J) (Koch et al., 2018).
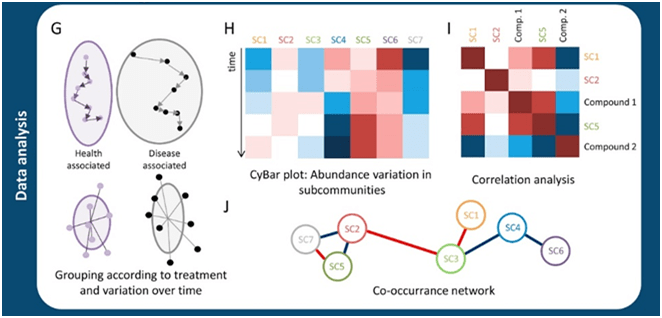
Figura 4. Flujo de trajo para el análisis de datos de microbiota adquiridos por citometría de flujo. G) Agrupación de acuerdo a los tratamiento y variaciones en el tiempo; H) CyBar plot: abundancia de las subcomunidades; I) Análisis de correlación; J) Red de co-ocurrencia o red semántica. Figura modificada de Koch et al., 2018.
DIFERENCIAS EN LA HUELLA CITOMÉTRICA DE LA MICROBIOTA DE RATONES SANOS vs COLITIS
Zimmermann J. et al., (2016) detectaron diferencias radicales en la composición de la microbiota fecal durante la colitis murina empleando en su estudio la citometría de flujo. Las poblaciones que diferían entre la huella citométrica de los ratones sanos y los ratones con colitis se aislaron mediante sorter, para la posterior extracción de su DNA y validación de las diferencias filogénicas mediante la secuenciación del gen ADNr 16S.
En la investigación se emplearon pooles de 12 ratones sanos y 11 ratones enfermos. A estos últimos se les induce la colitis mediante la transferencia de células T. Los ratones fueron sacrificados a las 2-3 semanas de la transferencia con signos de diarrea y pérdida de peso, en el caso de los enfermos. Las bacterias fecales se aislaron, fijaron con PFA 2% y permeabilizaron con etanol al 70% para mantenerlas a -20ºC hasta futuros análisis. Finalmente, se tiñeron con DAPI durante 3h en oscuridad.
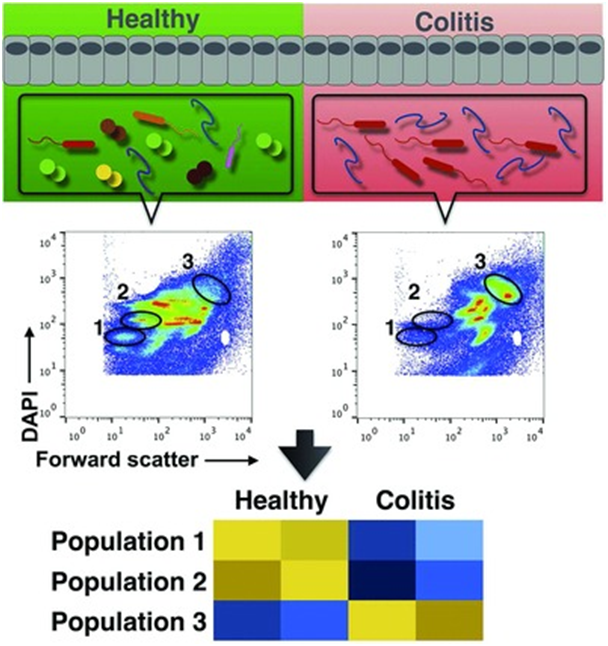
Figura 5. Comparativa de las huellas citométricas detectadas en ratones sanos frente a ratones con colitis. Utilizando los parámetros de dispersión frontal (FSC, eje-x) y el contenido de ADN (DAPI, eje-y) en las bacterias fecales, se detectaron poblaciones bacterianasque difieren en la microbiótica de ambos grupos (Zimmermann J., et al 2016).
Para el análisis de citometría de flujo de las diferentes muestras, se utilizó el flowCyBar, para el cual se realizó una plantilla de gates compuesta por todas las muestras del estudio. En la figura 5 podemos observar como hay dos poblaciones (gates 1 y 2) cuya frecuencia disminuye considerablemente durante la inflamación del colon; mientras que, existe una tercera población (gate 3), que aumenta significativamente en comparación con la microbiota de los mismos ratones antes de la inducción de la colitis. La disbiosis detectada mediante el análisis de las huellas citométricas, fue validada mediante la separación de los 3 gates por citometría y la secuenciación del gen ADNr 16s en cada uno de ellos.
CONCLUSIÓN
La citometría de flujo de alta resolución se presenta como un método innovador, rápido y económico para la identificación y la cuantificación de especies bacterianas de manera individual. Además, ofrece la oportunidad única de aislar poblaciones bacterianas definidas para análisis moleculares y determinación de funciones adicionales. Todo ello, está contribuyendo a conocer con mayor profundidad las características individuales de la microbiota intestinal y sus modificaciones por factores externos.
En definitiva, la citometría de flujo es una potente herramienta que permite establecer el perfil de ecosistemas bacterianos complejos mediante el análisis de la huella citométrica resultando una herramienta clave en clínica para vincular los cambios dinámicos de la microbiota intestinal con el diagnóstico y pronóstico de enfermedades.
AUTORA
Ángela Marquina Rodríguez
Técnico Titulado Superior
Hospital Nacional de Parapléjicos.
Toledo, España.
Referencias
- Loh, J. S., Mak, W. Q., Tan, L. K. S., Ng, C. X., Chan, H. H., Yeow, S. H., Foo, J. B., Ong, Y. S., How, C. W., & Khaw, K. Y. (2024). Microbiota-gut-brain axis and its therapeutic applications in neurodegenerative diseases. Signal transduction and targeted therapy, 9(1), 37. https://doi.org/10.1038/s41392-024-01743-1
- Koch, C., Günther, S., Desta, A. F., Hübschmann, T., & Müller, S. (2013). Cytometric fingerprinting for analyzing microbial intracommunity structure variation and identifying subcommunity function. Nature protocols, 8(1), 190–202. https://doi.org/10.1038/nprot.2012.149
- Koch, C., Harnisch, F., Schröder, U., & Müller, S. (2014). Cytometric fingerprints: evaluation of new tools for analyzing microbial community dynamics. Frontiers in microbiology, 5, 273. https://doi.org/10.3389/fmicb.2014.00273
- Zimmermann, J., Hübschmann, T., Schattenberg, F., Schumann, J., Durek, P., Riedel, R., Friedrich, M., Glauben, R., Siegmund, B., Radbruch, A., Müller, S., & Chang, H. D. (2016). High-resolution microbiota flow cytometry reveals dynamic colitis-associated changes in fecal bacterial composition. European journal of immunology, 46(5), 1300–1303. https://doi.org/10.1002/eji.201646297
- Koch, C., & Müller, S. (2018). Personalized microbiome dynamics – Cytometric fingerprints for routine diagnostics. Molecular aspects of medicine, 59, 123–134. https://doi.org/10.1016/j.mam.2017.06.005
- Schmiester, M., Maier, R., Riedel, R., Durek, P., Frentsch, M., Kolling, S., Mashreghi, M. F., Jenq, R., Zhang, L., Peterson, C. B., Bullinger, L., Chang, H. D., & Na, I. K. (2022). Flow cytometry can reliably capture gut microbial composition in healthy adults as well as dysbiosis dynamics in patients with aggressive B-cell non-Hodgkin lymphoma. Gut microbes, 14(1), 2081475. https://doi.org/10.1080/19490976.2022.2081475.
- Lambrecht, J., Schattenberg, F., Harms, H., & Mueller, S. (2018). Characterizing Microbiome Dynamics – Flow Cytometry Based Workflows from Pure Cultures to Natural Communities. Journal of visualized experiments : JoVE, (137), 58033. https://doi.org/10.3791/58033
Para más información, dudas o solicitud de presupuestos puedes contactarnos en: citometria.hnp@sescam.jccm.es
Si quieres conocer más sobre nosotros: Citometría HNP
Síguenos en:
www.linkedin.com/in/servicios-de-apoyo-a-la-investigación-sais-hnp