En esta newsletter queremos hablaros un poco sobre una técnica analítica ampliamente utilizada, la espectrometría de masas por imágenes (IMS).
Comprender la biodistribución, el metabolismo y la acumulación de fármacos en el cuerpo es un elemento fundamental en investigacióny enel desarrollo farmacéutico.LaIMSes una tecnología de vanguardia que puede proporcionar una amplia información molecular/química sobre biomoléculas, complementaria a la información que se obtiene con otros métodos de análisis espacial de muestras biológicas (Hematoxilina/ eosina (H&E), inmunohistoquímica (IHC)). Su objetivo es detectar los componentes moleculares (metabolitos, péptidos, proteínas) de la muestra, que pueden correlacionarse con las características histológicas y, en última instancia, proporcionar un vínculo molecular fenotípico con la compleja biología de una patología.
Introducción.
Desde el final de los años 90, cuando se empezó a utilizar la espectrometría de masas MALDIpara analizar secciones de tejidos de mamíferos y producir imágenes iónicas de péptidos y proteínas,se ha confirmado la habilidad de cartografiar moléculas particulares en coordenadas bidimensionales, dando lugar a la creación de imágenes moleculares.
Entre las diversas técnicas de ionización, MALDI IMS es líder en el análisis de distribuciones moleculares en diversos tejidos y diferentes enfermedades. MALDI IMS es capaz de cartografiar biomoléculas de interés con alta resolución espacial (∼10 μm) y alta sensibilidad.
Flujo de trabajo para la adquisición de imágenes por espectrometría de masas.
¿Qué tipo de muestras puedo utilizar?
Pueden utilizarse muestras de tejido fresco congelado, pero también muestras de tejido fijadas en formalina e incluidas en parafina (FFPE), incluidos los microarrays de tejidos (TMA).
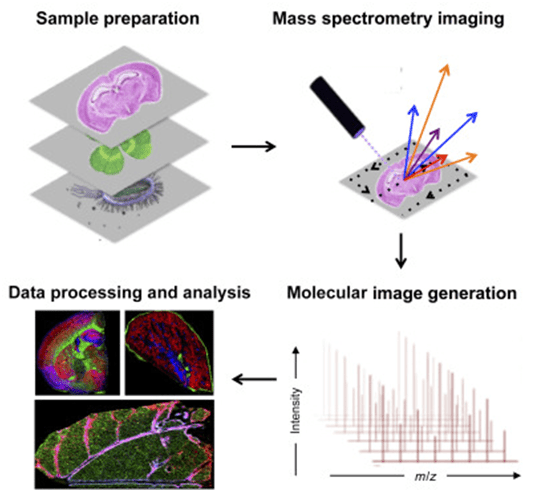
Figura 1 Visión general del flujo de trabajo en la obtención de imágenes por espectrometría de masas.[8]
La preparación y conservación de las muestras son los primeros pasos críticos para obtener imágenes iónicas de excelente calidad durante un experimento MALDI IMS. La estandarización de los procedimientos de preparación es necesaria para obtener resultados reproducibles, especialmente cuando se manejan grandes conjuntos de muestras o en un entorno clínico.
Respecto a la preparación de muestra, no existe un método que se adapte a todos los tejidos y analitos. Cada paso del proceso dentro del flujo de trabajo debe evaluarse y optimizarse en función de los tejidos, las hipótesis y los objetivos de investigación.
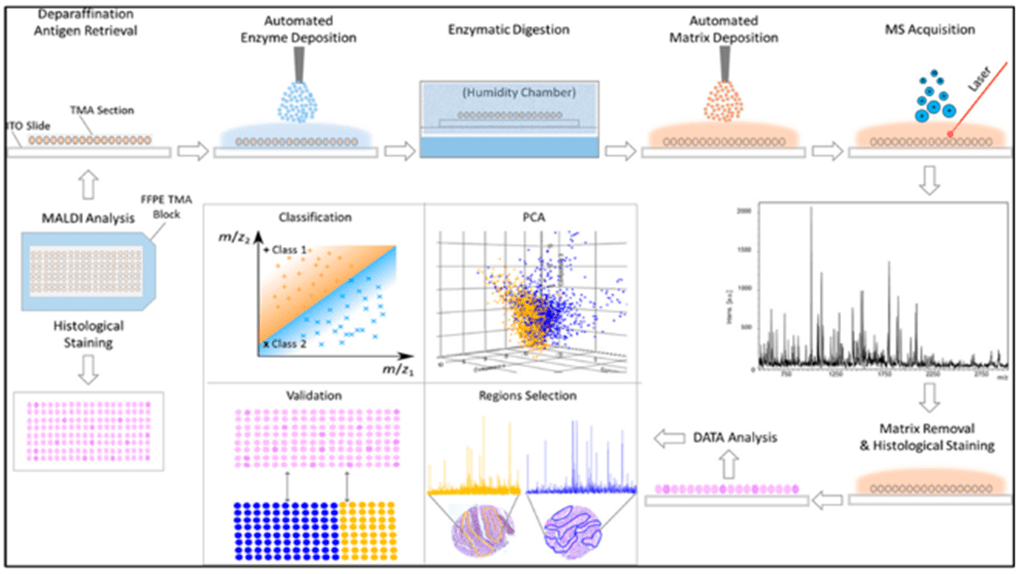
Figura 2 Flujo de trabajo para muestras FFPE TMA para detección de proteínas
El flujo de trabajo de MALDI IMS incluye una serie de pasos distintos que comienzan con el seccionamiento del tejido, que puede variar entre 5 y 15 μm de grosor dependiendo del tejido. El lavado del tejido para eliminar sales y lípidos interferentes es vital para el análisis de proteínas. La elección de matrizse realizará en función del tipo de analito objeto de estudio, ya que el éxito del análisis depende de que la matriz elegida favorezca una ionización y fragmentación eficiente del mismo. A continuación, el espectrómetro de masas recoge de forma automatizada espectros de masas de ubicaciones únicas a lo largo de la superficie de la muestra, y cada ubicación de muestreo contiene un espectro de masas único. Los espectros de masas adquiridos se combinan y procesan mediante algoritmos de software para crear una imagen MS analizable.
Tras la adquisición por MS, puede retirarse la matriz y teñir la muestra restante. Las secciones de tejido teñidas se utilizan para definir áreas de interés.
Aplicaciones.
MALDI IMS combina una sensibilidad y selectividad sin precedentes gracias a la espectrometría de masas de alta resolución, que puede combinarse con la histología tradicional. Esta tecnología única ha ganado popularidad en los últimos años en diversos campos de investigación, como la investigación médica, las plantas, los hongos y las bacterias.
Investigación médica:
Tejidos: MALDI IMS se ha aplicado con éxito a varios tejidos diferentes y en la investigación médica de diversas enfermedades. Uno de los campos en los que más se ha investigado es el cáncer, pero en los últimos años se postula como herramienta emergente en neurología para comprender mejor la patología celular de la enfermedad y/o su gravedad[3][4].
Aplicada a lesión medular se han detectado y localizado de forma específica acilcarnitinas de cadena larga a lo largo de los márgenes de la lesión a los 3 días de inducir la lesión y manteniéndose entre 7 y 10 días. [5].
En estudios de enfermedades relacionadas con el dolor, se ha investigado la distribución de neuropéptidos en médula (predominantemente localizados en la médula espinal dorsal) relacionados con el dolor. [6].
Otro ejemplo lo encontramos en el estudio de desmielinización de la médula espinal murina[7] en el que determinaron los cambios lipídicos que ocurren principalmente dentro de las lesiones en la desmielinización primaria.
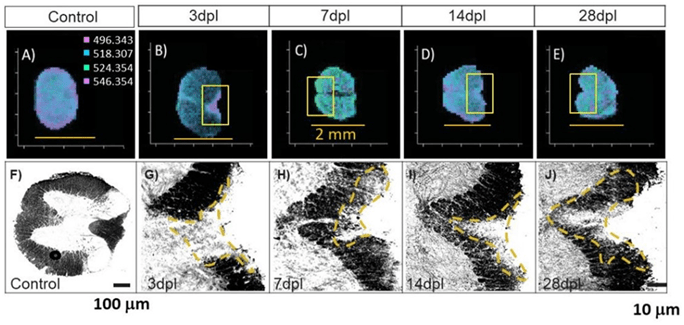
Figura 3 MALDI MSI de (A) secciones de médula espinal lesionadas, (B) 3 dpl, (C) 7 dpl, (D) 14 dpl y (E) 28 dpl (barras de escala = 2 mm) y el tejido teñido correspondiente de secciones de médula espinal (F) no lesionadas, (G) 3 dpl, (H) 7 dpl, (I) 14 dpl y (J) 28 dpl. Las inserciones en caja de los paneles B-E corresponden a las imágenes teñidas G-J.
Células:Además de los tejidos, la obtención de imágenes celulares por MALDI IMS ha ido ganado popularidad; se ha trabajado mucho en esferoides y se está trabajando mucho en la obtención de imágenes unicelulares.
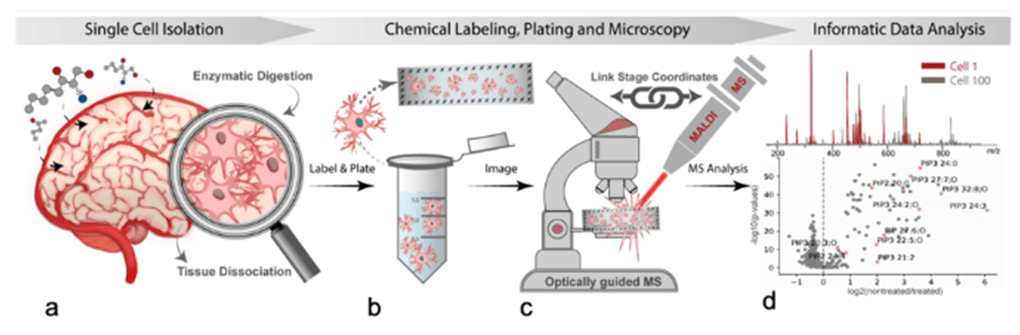
Figura 4 Flujo de trabajo de células individuales por espectrometría de masas MALDI guiada por imágenes. (a) Una región tisular aislada se somete a digestión enzimática para eliminar la matriz extracelular y los componentes conectivos, liberando las células individuales en la solución. (b) A continuación, las células se tiñen con una tinción nuclear y la solución de células individuales se extiende a través de un portaobjetos de microscopía que tiene marcadores fiduciales grabados alrededor del perímetro. (c) Se realiza una microscopía de campo claro y fluorescencia para determinar la ubicación relativa de las células en el portaobjetos. Se registran las coordenadas de píxel de los marcadores fiduciales y se inserta el portaobjetos en el espectrómetro de masas MALDI. Utilizando el punto de mira de la cámara MALDI, se encuentran los mismos puntos fiduciales y se registran sus coordenadas. A continuación se realiza el registro de la imagen utilizando los marcadores fiduciales para traducir las coordenadas de píxel de las células individuales a las coordenadas físicas de la platina del espectrómetro de masas. (d) Espectrometría de masas MALDI de alto rendimiento y análisis de estilo informático en el que cada adquisición MALDI representa una única célula. Imagen inédita, cortesía del Dr. Jonathan Sweedler y Dan Castro de la Universidad de Illinois Urbana-Champaign.
Ventajas y Desventajas
Ventajas.
- Capacidad para detectar, cuantificar relativamente y mapear moléculas pequeñas (<2kDa) y los metabolitos dentro de una muestra.
- Alta resolución espacial y especificidad molecular sin necesidad de marcadores químicos, procedimientos de tinción y sondas moleculares.
- Complemento a las técnicas de histología.
Desventajas.
Las principales desventajas que presenta derivan del proceso de preparación de la muestra.
- Supresión de iones: ocurre cuando las naturalezas químicamente distintas de cada metabolito perjudican la detección general. Los analitos más abundantes se ionizan selectivamente sobre los menos abundantes.
- Degradación de la muestra, que puede ocurrir cuando el tejido se descongela durante la preparación de la muestra.Ciertos analitos no son estables a temperatura ambiente y pueden degradarse por completo, lo que lleva a una interpretación errónea de los datos.
- La aplicación no uniforme de la matriz puede producir artefactos.
- No existe una estandarización de la preparación de la muestra.

Los métodos alternativos de ionización ya han demostrado ser eficaces para eludir algunos de estos problemas. Por ejemplo, la MS de iones secundarios minimiza los problemas con las inconsistencias de la matriz y la difusión del analito al no requerir la aplicación de la matriz en absoluto.
Conclusiones
MALDI IMS sigue avanzando en la comprensión científica de las enfermedades al proporcionar información espacial sobre los analitos dentro de los sistemas biológicos. Comprender dónde se encuentran las biomoléculas en relación con la patología y en relación con otras biomoléculas permite a los investigadores un conocimiento profundo del sistema. En las últimas décadas, se ha puesto énfasis en el avance de esta técnica para encontrar alternativas que aumenten la sensibilidad de la detección, mejoras en la instrumentación y una mayor comprensión en la preparación de muestras. Estos avances han llevado a reforzar el uso de MSI para el análisis de terapias y biomoléculas.
Bibliografía
[1] J. L. Moore and G. Charkoftaki, “A Guide to MALDI Imaging Mass Spectrometry for Tissues,” J. Proteome Res., vol. 22, no. 11, pp. 3401–3417, 2023, doi: 10.1021/acs.jproteome.3c00167.
[2] J. H. Holbrook, G. E. Kemper, and A. B. Hummon, “Quantitative mass spectrometry imaging: therapeutics & biomolecules,” Chem. Commun., pp. 2137–2151, 2024, doi: 10.1039/d3cc05988j.
[3] K. Chen, D. Baluya, M. Tosun, F. Li, and M. Maletic-Savatic, “Imaging mass spectrometry: A new tool to assess molecular underpinnings of neurodegeneration,” Metabolites, vol. 9, no. 7. 2019. doi: 10.3390/metabo9070135.
[4] L. K. Schnackenberg, D. A. Thorn, D. Barnette, and E. E. Jones, “MALDI imaging mass spectrometry: an emerging tool in neurology,” Metab. Brain Dis., vol. 37, no. 1, pp. 105–121, 2022, doi: 10.1007/s11011-021-00797-2.
[5] J. Quanico et al., “3D MALDI mass spectrometry imaging reveals specific localization of long-chain acylcarnitines within a 10-day time window of spinal cord injury,” Sci. Rep., vol. 8, no. 1, p. 16083, Dec. 2018, doi: 10.1038/S41598-018-34518-0.
[6] P. Sui et al., “Neuropeptide imaging in rat spinal cord with MALDI-TOF MS: Method development for the application in pain-related disease studies,” http://dx.doi.org/10.1177/1469066717703272, vol. 23, no. 3, pp. 105–115, May 2017, doi: 10.1177/1469066717703272.
[7] E. R. Sekera, D. Saraswat, K. J. Zemaitis, F. J. Sim, and T. D. Wood, “MALDI Mass Spectrometry Imaging in a Primary Demyelination Model of Murine Spinal Cord,” J. Am. Soc. Mass Spectrom., vol. 31, no. 12, pp. 2462–2468, Dec. 2020, doi: 10.1021/JASMS.0C00187/SUPPL_FILE/JS0C00187_SI_001.PDF.
[8] M. de Raad, T. R. Northen, and B. P. Bowen, “Analysis and Interpretation of Mass Spectrometry Imaging Datasets,” Compr. Anal. Chem., vol. 82, pp. 369–386, Jan. 2018, doi: 10.1016/BS.COAC.2018.06.006.
AUTOR
Gemma Barroso García, MSc.
Responsble SAI-Proteómica.
Hospital Nacional de Parapléjicos.
Toledo, España.
Para más información, dudas o solicitud de presupuestos puedes contactarnos en: unidadproteomica.hnp@sescam.jccm.es
Si quieres conocer más sobre nosotros: https://hnparaplejicos.sanidad.castillalamancha.es/profesionales/investigacion/servicios-apoyo/proteomicahttps://saishnp.com/
Síguenos en:
www.linkedin.com/in/servicios-de-apoyo-a-la-investigación-sais-hnp