En las dos Newsletter anteriores de esta serie, hemos descrito en detalle el microscopio IX83 de Olympus (Newsletter 1, de Enero 2022) y además hemos introducido los conceptos básicos de la inteligencia artificial directamente relacionados con el análisis de la imagen (Newsletter 2, de Enero 2023). En esta tercera y última Newsletter de la serie explicaremos tres ejemplos con los que hemos trabajado utilizando la IA para analizar las imágenes capturadas. Con los tres ejemplos que describimos podréis haceros una idea de la versatilidad que tenemos a la hora de analizar imágenes mediante IA.
La Newsletter está estructurada en los diferentes pasos a seguir para realizar el proceso de Deep learning, que podéis ver resumidos en el esquema de la figura 1. Cada apartado de la Newsletter es uno de los pasos del esquema (figura1) esto permite seguir el proceso más fácilmente (pasos 1, paso 2, etc), y dentro de cada paso se explican los 3 ejemplos diferentes que estamos describiendo.
Nota: algunos pasos son simultáneos en el tiempo y se explican en un orden no consecutivo para entender mejor el proceso.
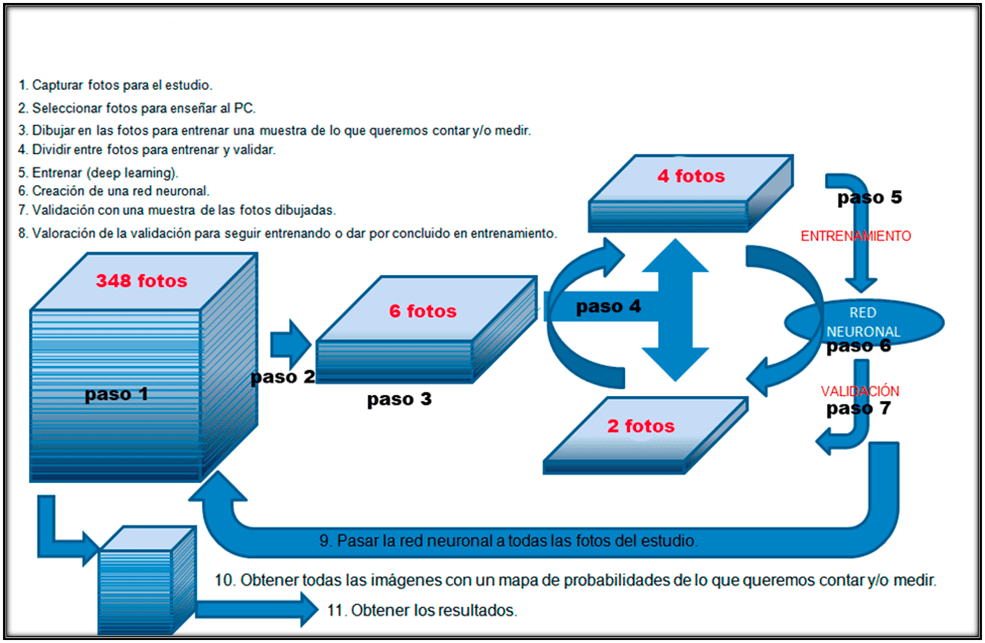
Figura 1. Esquema de los diferentes pasos en la utilización del Deep learning para crear redes neuronales para el análisis de imágenes mediante Inteligencia Artificial.
Los tres ejemplos que iremos explicando paso a paso son los siguientes:
EJEMPLO 1 Fotos fibras musculares. Fotos capturadas en un microscopio diferente al que se utilizara para crear la red neuronal. Identificación de fibras de musculo liso oxidativas y no oxidativas (tipo 1 y tipo 2) en diferentes animales con diferentes tratamientos.
EJEMPLO 2 Fotos cortes de medula para contar neuronas. Fotos capturadas con el mismo equipo (microscopio IX83 de Olympus) y el mismo y software (Cellsense) con el que se entrena y crea la red neuronal con la que se analizarán las fotos capturadas. Identificación de neuronas en medula.
EJEMPLO 3 Fotos de células en cultivo para contar núcleos. Fotos capturadas con el mismo equipo (microscopio IX83 de Olympus) pero diferente software (ScanR) con el que se entrena y crea la red neuronal con la que se analizarán las fotos capturadas. Para identificación de núcleos raros (cuando los núcleos son poco convencionales debido a tratamientos especiales) para contar células en HCS (High Content Analisis).
A. CAPTURA
PASO 1. Captura de las fotos.
EJEMPLO 1. Fotos fibras musculares
Se utilizó el equipo de estereología (BX61), pero se podría haber utilizado cualquier microscopio de los que tenemos en el servicio, con una cámara color (DP-70) y un objetivo 10X, intentando hacer un máximo de 10 fotos por animal.

Figura 2. Algunas de las fotos capturadas de fibras musculares (Ejemplo 1)
EJEMPLO 2. Fotos cortes de médula espinal para contar neuronas. (Este sería el ejemplo más común de captura de fotos, el más automatizado y el que se realiza con el mismo microscopio donde se analizará).
1. Decisión las características del experimento de captura, principalmente tipo de iluminación (transmitida/ fluorescencia) potencia de la luz, filtros de fluorescencia, tiempo de exposición, offset etc. (Fijar Settings)
2. Determinar la ruta donde se almacenaran las fotos que capturaremos de forma automática y además el nombre* con el que se guardaran cada una de ellas.
Nombre*; nombre del experimento (general para todas las fotos)_separador_ posición de grupo (especifico para cada porta)_ separador_ contador (un número, especifico de cada corte o ROI)
3. A continuación se obtiene un mapa o vista general a baja resolución (4X) de la zona de la platina donde colocamos la muestra. Este mapa o vista general (overview) nos permitirá navegar por la muestra que tenemos en la platina. Con esto obtenemos una imagen a baja resolución de los portaobjetos colocados en el adaptador múltiple de la platina (de 1 a 4 porta objetos).
4. Determinar sobre el mapa o la vista general la ROI o zona de interés* que queremos fotografiar con mayor resolución (ver ROIs en azul figura 3).
5. Agrupamos cada grupo de cortes en otra ROI más grande (rojo, verde figura 3) que engloba todos los cortes de un mismo grupo (porta, tratamiento, individuo…), será lo que en el nombre definitivo de la foto se denomina posición de grupo (ver nombre*).

6. Fijación del mapa de foco*.Determinar tres puntos de foco por cada corte (ROI), para crear un plano de foco. De esta, durante la captura, el microscopio enfocará de manera automática a lo largo de la muestra que va capturando. Si la región a fotografiar está compuesta por 4 o menos campos podemos enfocar cada vez que se dibuja una ROI (azul figura 3) y no será necesario hacer el plano de foco.
*Mapa de foco; Fijar de 3 puntos de foco por cada región (ROI) a fotografiar, con tres puntos de foco definimos completamente el plano y por tanto se irá ajustando el foco a lo largo del plano a fotografiar en relación a los tres puntos de foco.
7. Capturamos a mayor resolución (10X en este ejemplo) las imágenes en mosaico de las regiones definidas anteriormente en el overview o mapa. En otros ejemplos se ha utilizado el objetivo 20X o el 40X (pero en este caso haciendo Z-stack y un proceso de EFI “Extended focus imaging”, para tener todos los planos en uno solo completamente enfocado).
EJEMPLO 3. Fotos capturadas con el software ScanR para realizar HCS. (Ver Newsletter Jose Angel Rodriguez el 28 de Junio 2021)
En este ejemplo se diseña un experimento de captura de fotos en placas de cultivo utilizando el programa ScanR de Olympus. Este programa también lo tenemos en el microscopio IX83 motorizado de Olympus y está diseñado principalmente para hacer HCA (High Content Analysis), normalmente en placa de cultivo y captura de forma automática multitud de fotos de cada pocillo de la placa de cultivo.
El ScanR está compuesto por dos Softwares, el programa de Captura (para adaptar la captura a cualquier placa de cultivo) y el programa de Análisis (que es parecido a los programas de citometría, para analizar poblaciones de eventos). Con el ScanR podemos entrenar el equipo para crear redes neuronales que nos permitan analizar las imágenes aplicando inteligencia artificial (Newsletter de Jose Angel Rodriguez el 28 de Junio 2021). Pero en este caso, la red neuronal utilizada para el análisis de las fotos capturadas con ScanR la crearemos con el Software Cellsens. Esto lo hacemos puesto que el software Cellsens es más flexible a la hora de etiquetar las fotografías a analizar durante el entrenamiento (ver paso 3). Pese a que la captura e incluso el análisis se realizaran con otro programa (ScanR) podemos utilizar las fotos capturadas para crear una red neuronal en el programa Cellsens.
En este caso crearemos una red neuronal que identifica núcleos complejos (poco convencionales) y nucleos normales, para identificar células independientes en el proceso de HCA del ScnaR (ver Newsletter de Jose Angel Rodriguez el 28 de Junio 2021). Es decir, después de capturar las imágenes de los diferentes pocillos de la placa con ScanR de captura, utilizaremos algunas fotos para entrenar una red neuronal con Cellsens. Una vez tengamos la red neuronal, la utilizaremos en ScanR de análisis, para analizar y agrupar en poblaciones las células con núcleos raros de las fotografías capturadas en los diferentes pocillos de la placa de cultivos.
RESUMEN CAPTURA
Terminado el proceso de captura (Paso 1) tenemos multitud de fotos para analizar. Las fotografías que tenemos son:
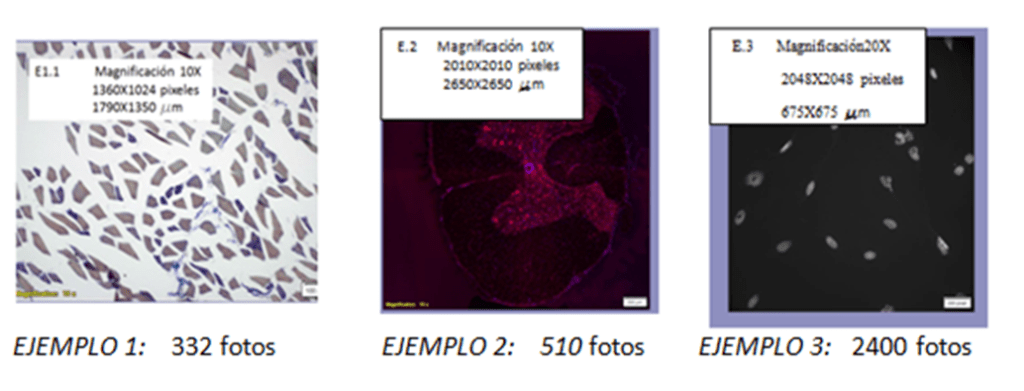
Figura 3. Tipos y número de de fotos capturadas para cada tipo de ejemplo.
B. ENTRENAMIENTO
PASO·2. Selección de las fotos para entrenar
Una vez capturadas las fotos de cada ejemplo se realiza una selección al azar de entre un 1% a un 10% de las fotos capturadas (dependiendo del número total de fotos y de la complejidad de lo que queremos identificar), para realizar el entrenamiento de la red neuronal.
Ejemplo 1. De las fibras musculares se seleccionó un 10% de las fotos, en este caso seleccionamos un alto porcentaje de fotos para entrenar, debido a que el entrenamiento es un tanto complejo, puesto que debemos distinguir entre dos tipos de fibras y lo que no es fibra. Además no tenemos muchas fotos, por tanto un 10% no es un número excesivo para entrenar.
Se seleccionaron 33 fotos para entrenar de las 332 adquiridas.
Ejemplo 2. De los cortes de medula para la identificación de neuronas, se seleccionó un 5% de las fotos, en este caso las fotos son muchos más grandes y con más ejemplos de neuronas en cada una, además tenemos más fotos totales. En este ejemplo el problema es asegurarnos de introducir en la selección, fotos con lesión donde la zona de inflamación puede producir errores de contaje de neuronas, esto quiere decir que en este caso no debemos seleccionar todas las fotos al azar.
Se seleccionaron 25 fotos para entrenar de las 510 adquiridas.
Ejemplo 3. De las fotos de núcleos raros seleccionamos sólo un 2 %. En este caso un 2% es un número elevado de fotos, puesto que partimos de 2400 fotos (se capturaron de forma automática con ScnaR). Aquí tenemos que asegurarnos de introducir fotos de pocillos sin tratamiento (con núcleos normales) y otras con núcleos raros (fotos de pocillos con tratamiento), para entrenar al PC en la identificación tanto de núcleos normales como lobulados o raros.
Se seleccionaron 48 fotos de entrenamiento (20 con núcleos normales y 28 con núcleos raros) de las 2400 adquiridas.
PASO 3. Etiquetado de imágenes. Dibujando sobre las fotos seleccionadas las mascaras que distingan las diferentes zonas o estructuras a identificar para el entrenamiento.
Una vez seleccionadas las fotos para el entrenamiento deben ser etiquetadas para identificar los objetos de interés para nuestro estudio. Para ello utilizamos la interface (pestaña o layout) de “Count and measure” del programa Cellsense (figura 4, 5 y 6).
EJEMPLO 1
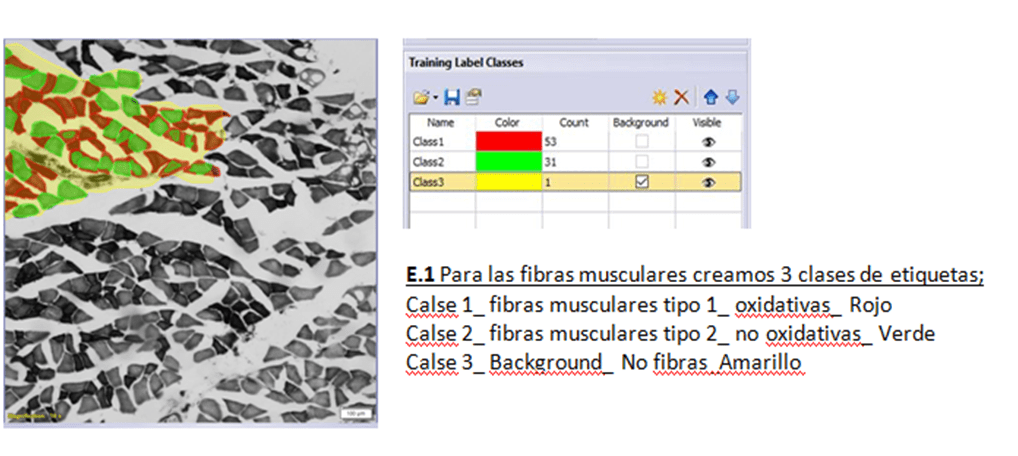
Figura 4. Etiquetas de entrenamiento para el ejemplo 1
EJEMPLO 2
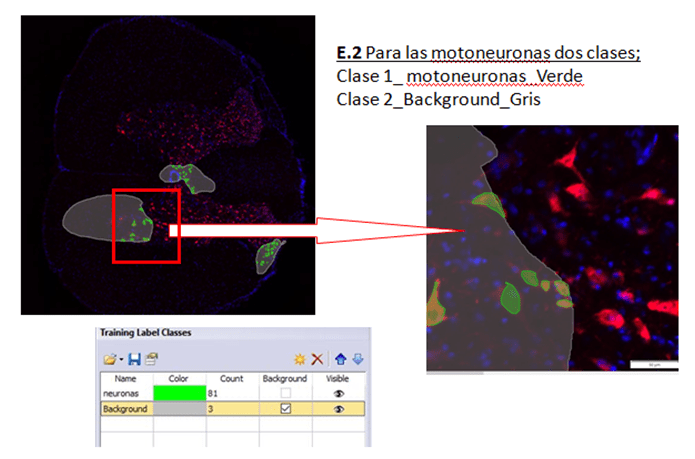
Figura 5. Etiquetas de entrenamiento para el ejemplo 2
EJEMPLO 3
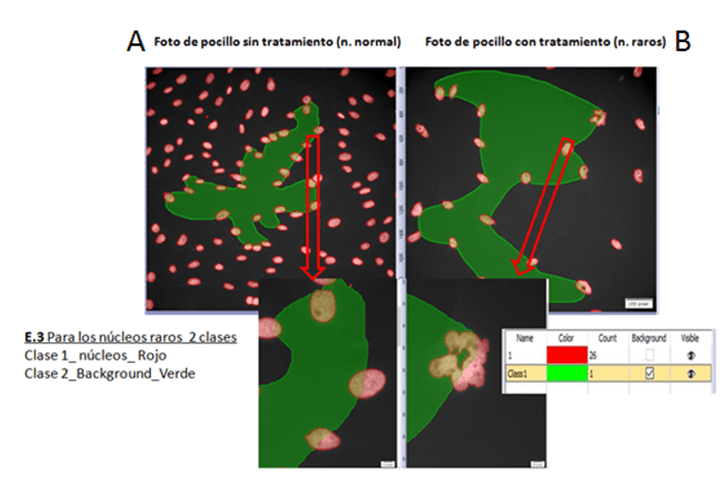
Figura 6. Etiquetas de entrenamiento para el ejemplo 3
Utilizando las etiquetas que se muestran en las figuras 4, 5 y 6, se dibujan manualmente por el investigador las fotos seleccionadas para entrenar. No es necesario etiquetar el 100 % de la foto y siempre es necesario crear la etiqueta de Background, donde se identifica todo lo que no interesa y que rodea a la zona etiquetada.
En el caso del ejemplo 3 el etiquetado fue semiautomático, para dibujar las máscaras que identifican los núcleos en las fotos para el entrenamiento, se recurrió a los pasos clásicos de segmentación de la imagen, mediante un ajuste de Threshold, un watershed y una binarización de forma automática. Tras obtener el resultado de segmentación, las fotos fueron editadas. Esta edición fue manual y consistió en ir uniendo zonas que el watershed había separado y separando zonas que el watershed no ha identificado, debido a la complejidad de los núcleos raros que tenían formas muy heterogéneas (ver Figura6 B). Esta parte de edición la hacemos de manera similar y con las mismas herramientas que utilizamos para dibujar las mascaras en los ejemplos 1 y 2.
C. ENTRENAMIENTO
PASO 5. Entrenamiento (Deep Learning). Nota: Pasos 4 y 5 son simultáneos en el tiempo (ver figura 1).
Para realizar el entrenamiento con el software Cellsens utilizamos el Layout o interface de Deep learning del programa (figura 7). En esta interface del programa añadimos las fotos etiquetadas o dibujadas en el paso anterior. Estas fotos y sus máscaras servirán para que el programa diseñe una red neuronal aprendiendo de las fotos marcadas o etiquetadas mediante deep learning (Newsletter 1, de Enero de 2022 o Newsletter 2, de Enero 2023).
Uno de los puntos importantes del entrenamiento es decidir el tipo de Deep learning que realizará el equipo y el número de iteraciones que realizará. Cada tipo de deep learning utiliza unos algoritmos diferentes, en los tres ejemplos que nos ocupan, hemos utilizado el estándar (Standard Network). Por último, se fijan el número de iteraciones que realizara el entrenamiento. Las iteraciones son repeticiones del proceso de entrenamiento hasta obtener el resultado deseado, comenzamos con 250 k pero podemos llegar a más de un millón.
Nota: Standard Network, utiliza una arquitectura U-Net con 32 mapas de características en la primera capa de convolución. La red aplica rotaciones de 90° y duplicación de las imágenes etiquetadas para aumentar los datos de entrenamiento.
PASO 4. Selección de las imágenes de validación. Nota: Pasos 4 y 5 son simultáneos en el tiempo (ver figura 1).
Este paso es automático, y lo realiza el propio programa una vez comience el proceso de entrenamiento. En el paso anterior son cargadas las fotos etiquetadas en la ventana de entrenamiento. Y de todas las fotos que fueron etiquetadas manualmente y cargadas en la interface de entrenamiento, el programa selecciona de forma automática entre un 1 y el 10 % de estas fotos, y las utilizara para validar, sin ser utilizadas para el entrenamiento. Es decir, de las fotos etiquetadas manualmente, el programa separa un pequeño porcentaje de ellas que serán utilizadas para validar y no para entrenar.
E1. Fotos adquiridas: 332. Fotos de entrenamiento: 33. Fotos de validación: 3
E2. Fotos adquiridas: 510. Fotos de entrenamiento: 25. Fotos de validación: 3
E3. Fotos adquiridas: 2400. Fotos de entrenamiento: 48. Fotos de validación: 5
D. VALIDACIÓN.
PASO 7 y 8. Validación y valoración de la Validación. Nota: Pasos 6, 7 y 8 son simultáneos en el tiempo (ver figura 1).
Cuando comienza el entrenamiento, el programa muestra una grafica en la que se representan valores de similaridad frente a las iteraciones realizadas durante el entrenamiento (figura7 C), cuantas más iteraciones se produzcan mayor será la similaridad, hasta un punto en el que el valor se mantiene constante o incluso empieza a decaer. Pero, ¿cómo podemos definir el valor de similaridad? Pues bien, el valor de similaridad es, como se parece la predicción hecha por el programa aplicando la red neuronal creada hasta ese momento (después de realizar unas iteraciones concretas) con las etiquetas de las fotos de validación que hemos dibujado manualmente en las fotografías seleccionadas para validar (es decir, en las fotografías etiquetadas y reservadas para validación sin ser utilizadas para crear la red neuronal). Los valores de similaridad son un porcentaje y se representan de 0 a 1. Es importante resaltar que las fotos de validación no se utilizaron en el entrenamiento para crear la red neuronal. De manera que pese a estar etiquetadas el programa no las ha utilizado para aprender. Además cada cierto número de iteraciones el programa ira fijando automáticamente unos check-point, con los valores de similaridad específicos a esas iteraciones.
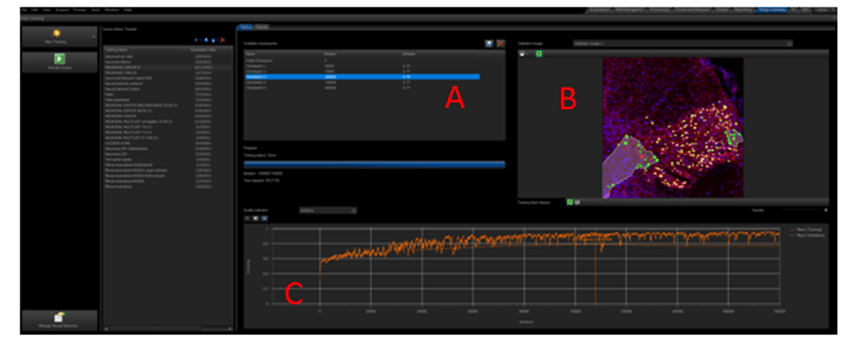
Figura 7. Layout o interface de Deep learning en el software Cellsens. Donde vemos los diferentes check point (A), una imagen de validación (B) y la grafica de similitud con respecto a las iteraciones (C).
Y aquí tenemos la grafica de validación (Figura 7C) y la imagen de validación (Figura 7B y Figura 8) con la predicción hecha sobre la imagen a las 110000 iteraciones o Check point 3 (Figura 7A).
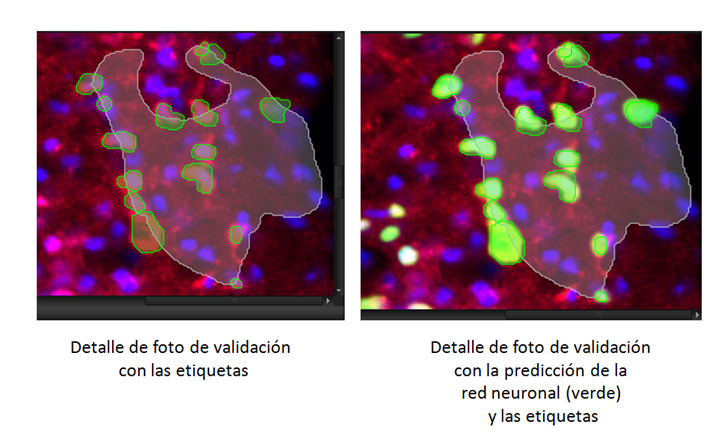
Figura 8. Detalle de la foto de validación al comienzo del entrenamiento y en el tercer check point (110000 iteraciones) con un valor alto de similaridad.
Cuando los valores de validación o similaridad son altos (cercanos al 0.8, es decir un 80 % de similaridad), es porque la predicción de la red neuronal creada hasta el momento considera que se aproxima mucho a las etiquetas que creamos manualmente. Existen diferentes medidas de validación, las más comunes son; el valor de similaridad, el Intersection over Union (IoU) o el coeficiente de similitud. Estos valores sirven para evaluar los algoritmos de Deep Learning al estimar como de bien una máscara predicha coincide con los datos reales del terreno. Es obvio, que cuanto mayor sea el valor de similaridad más se aproxima la predicción a la realidad. Así podemos ver que cerca de 110000 iteraciones del entrenamiento de las neuronas (E.2) tenemos unos valores de similaridad de más del 0.8 y que ese valor no aumenta por mas iteraciones que se realicen. Lo cual nos lleva a decidir que la red neuronal está terminada y podemos finalizar el proceso de entrenamiento fijando un check point a este número de iteraciones (ver figura 7).
E.1. En el caso del musculo programamos 150000 iteraciones y vimos que a partir de 30000 iteraciones los valores de similaridad eran altos llegando al máximo a las 50000 iteraciones con un Intersection over Union (IoU) de 0.79, a partir de aquí se estanco y a las 100000 iteraciones empezó a bajar el Intersection over Union (IoU).
E.3. En el caso de los núcleos se programaron 100000 iteraciones y en el primer check point de 20000 iteraciones el valor de similaridad ya era del 0.94.
E. RED NEURONAL CREADA DURANTE EL ENTRENAMIENTO.
PASO 6. Obtención de la red neuronal Nota: Pasos 6, 7 y 8 son simultáneos en el tiempo (ver figura 1).
Durante todo el proceso de entrenamiento mediante Deep learning el programa ha ido gravando automáticamente unos puntos de comprobación o Check point (figura 7 A). Esto lo se ha hecho de forma automática cada cierto tiempo, o mejor dicho, cada cierto número de iteraciones. Estos check-point también han podido ser grabados bajo nuestra supervisión a ciertas iteraciones en las que nos pareciera que los valores de similaridad eran suficientemente altos.
Con todos estos puntos de comprobación o check-point decidimos cual es el mejor para nuestro propósito y lo guardamos como red neuronal.
Una vez que tenemos la red neuronal, podremos identificar por Inteligencia Artificial las fibras musculares tipo 1 o 2 (ejemplo 1), las neuronas (ejemplo 2) o los núcleos aunque estos sean raros (ejemplo 3).
F. OBTENCIÓN DEL MAPA DE PROBABILIDADES Y RESULTADOS.
PASO 9. Utilizar la red neuronal para identificar el propósito de nuestro estudio en el total de las fotos, obteniendo un mapa de probabilidad (PASO 10) y a partir del mapa de probabilidad los resultados (PASO 11).
Con la red neuronal establecida, se hace un análisis de todas las fotos del estudio aplicando la red neuronal a todas las fotos capturadas del experimento. Tras aplicar la red neuronal obtenemos una capa adicional sobre la foto capturada, que representa un mapa de probabilidades de identificación de las estructuras etiquetadas para el entrenamiento. El mapa de probabilidades representa la probabilidad de cada pixel de la foto a pertenecer a cada una de las clases etiquetadas en el entrenamiento A partir del mapa de probabilidad podemos obtener los resultados de diferentes formas.
E1. MÚSCULO. Utilizando la interface de “count and measure” del programa Cellsens creamos una macro con la pestaña “macro manager”. Esta macro consiste en aplicar la red neuronal obtenida en el paso anterior a todas y cada una de las fotos adquiridas, para poder hacer el proceso automáticamente (esta parte es común a los tres ejemplos).
Resultado de esta macro obtenemos una capa adicional en la imagen capturada que representa un mapa de probabilidades por cada pixel de la foto, que nos indica la probabilidad a pertenecer a cada una de las clases etiquetadas en el entrenamiento, es decir, a cada una de las fibras musculares o al background (figura 9 B).
Con el mapa de probabilidades por tipos de fibra creamos una segunda macro, para segmentar la imagen del mapa de probabilidad mediante un threshold. Determinamos un Threshold (umbral) para cada tipo de fibra y para el background y corremos la macro en las fotos con el mapa de probabilidades. De esta manera cuando corremos la macro obtendremos una imagen binarizada por tipo de fibra y background y una tabla con el porcentaje y área de cada tipo de fibra por foto (Figura 9C).

Figura 9. Detalle de una foto de fibras musculares (Ejemplo.1)( A), la misma foto con su mapa de probabilidad (B) y la misma imagen binarizada tras la segmentación que dará lugar a los resultados (C).
E2. NEURONAS. Pasamos la red neuronal obteniendo los mapas de probabilidad de cada una de las fotos. A partir de estas imágenes se obtuvieron los mapas de probabilidad como en el caso anterior. Se separaron las capas de los mapas de probabilidad y se analizaron en el programa libre Fiji. A partir de los mapas de probabilidad aislados, se segmentaron de forma muy sencilla y se contaron mediante la función de análisis de partículas de Fiji, obteniendo el número de neuronas por cada foto (corte).
E3. NUCLEOS RAROS. Una tercera opción que podemos hacer es enviar la red neuronal a el programa ScnaR y utilizarla para analizar las fotos capturadas para hacer HCA, en este caso para identificar los núcleos (sean normales o raros) con esta red neuronal. Utilizando la red neuronal para segmentar las imágenes capturadas con ScanR.
AUTOR
José Ángel Rodríguez Alfaro
Responsable del Servicio de Microscopía y Análisis de Imagen
Hospital Nacional de Parapléjicos.
Toledo, España.