En la actualidad, la microscopía es una disciplina fundamental en las investigaciones biomédicas. Con ella se obtienen imágenes que posteriormente se analizan para obtener resultados. Dentro de las distintas formas de extraer información de las imágenes, en los últimos años ha habido grandes avances en el campo de la inteligencia artificial. En concreto, para el análisis de este tipo de imágenes se están empleando cada vez con más frecuencia algoritmos conocidos como redes neuronales artificiales (ANNs), que se engloban dentro del deep learning (DL). Como con toda nueva herramienta, es necesario tener precaución y validar correctamente los resultados obtenidos. Desde la comunidad de analistas de imagen científica se han propuesto distintos parámetros de medición que permitan realizar esta validación(1). En esta newsletter nos vamos a basar en esa publicación para describir estos parámetros y, con la ayuda de los Colab Notebooks del proyecto ZeroCostDL4Mic(2,3), los aplicamos para evaluar distintos modelos de ANNs entrenados en el SMAI, comprobando cómo se relacionan con la calidad de la predicción obtenida al aplicarlos tanto para la detección de objetos como para la mejora de imágenes.
Todos los parámetros de medición que aquí se describen permiten la comparación de la predicción generada por modelos de DL a imágenes etiquetadas o de ground-truth. Cuál elegir depende del tipo de tarea realizada por el modelo de DL. En algunos casos puede haber varios parámetros de medición disponibles que se pueden usar juntos.
Parámetros de medición de similitud de imágenes (eliminación de ruido, restauración y traducción de imagen a imagen)
Se pueden utilizar varios parámetros de medición para evaluar la similitud de dos imágenes:
- El mapa RSE muestra la raíz cuadrada de la diferencia al cuadrado entre dos imágenes. En este caso, un RSE más pequeño es mejor. Una coincidencia perfecta entre la ground-truth y la predicción conducirá a un mapa RSE mostrando ceros en todas partes.
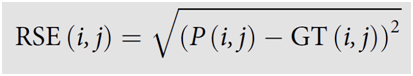
Donde P(i,j) es el valor de predicción en el píxel (i,j) y GT(i,j) es el valor del mismo píxel en la ground-truth. Habitualmente las imágenes se normalizan antes de la evaluación.
- La raíz cuadrada del error cuadrático medio normalizado (normalized root mean squared error NRMSE) nos da la diferencia media entre todos los píxeles de las imágenes comparados entre ellos.

Donde N es el número total de píxeles. Habitualmente las imágenes se normalizan antes de la evaluación.
- El coeficiente de correlación de Pearson (PCC) representa el grado de correlación lineal entre dos imágenes. Una correlación alta entre la predicción y la ground-truth produce un PCC cercano a 1.
- El SSIM evalúa si dos imágenes contienen las mismas estructuras basándose en los conceptos de contraste, luminancia y contenido estructural. Es un parámetro de medición normalizado y un SSIM de 1 indica una similitud perfecta entre las dos imágenes. Los mapas de SSIM se generan calculando el valor SSIM en cada píxel pero también considerando los píxeles circundantes. El mSSIM es el valor de SSIM calculado en toda la imagen.
- Pico de la relación señal-ruido (PSNR) estima las discrepancias entre dos imágenes con respecto al pico de la amplitud de la señal de la imagen de predicción. Suele calcularse en decibelios y cuanto mayor sea la puntuación, mejor será la concordancia.
En la siguiente figura podemos observar el resultado de usar estos parámetros en una imagen que ha sido tratada para reducir el ruido utilizando cuatro métodos distintos. En primer lugar, un desenfoque gaussiano de radio 1, un método tradicional de reducción de ruido (GAUSS). A continuación tenemos una imagen tratada con CARE(4), que entrena el modelo de ANN a partir de pares de imágenes con y sin ruido. Después tenemos el resultado de aplicar a la imagen «SOURCE» una ANN entrenada también con CARE, pero empleando pares de imágenes ruidosas para entrenar el modelo (Noise2Noise o N2N). Por último, usamos un modelo en el que en el entrenamiento partimos únicamente de imágenes ruidosas desemparejadas (Noise2Void(5) o N2V). Podéis ver más detalles de cómo se han generado estas imágenes y los modelos empleados en la newsletter de noviembre de 2020(6).

En la tabla de la figura podemos ver cómo con todos los métodos de reducción de ruido obtenemos valores mejores de los que se obtienen al comparar la imagen ruidosa (SOURCE) con la GT. Los mejores valores se obtienen con el empleo de CARE, seguidos por N2N. El desenfoque gaussiano produce unos valores solo un poco peores que N2N, pero son mejores que los obtenidos con nuestro modelo de N2V.
Parámetros de segmentación:
La segmentación de imágenes tiene como objetivo definir áreas de interés en una imagen en función de su identidad (elementos destacados frente al fondo es el más común). Una segmentación generalmente proporciona una imagen de máscara binaria donde los píxeles en el área segmentada tienen un valor de 1 (destacados) mientras que el resto de los píxeles tienen un valor de 0 (fondo).
- El parámetro de medición IoU es un método que se puede utilizar para cuantificar la superposición entre dos máscaras binarias. Por lo tanto, cuando se utiliza IoU para evaluar el rendimiento de un algoritmo de segmentación comparando el resultado con las máscaras de la ground-truth, cuantomás próximo a 1, mejor habrá funcionado el algoritmo.
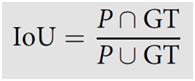
Donde ∪ representa la unión de dos imágenes binarias (número de píxeles destacados en cada una de las imágenes, ∩ representa la intersección de dos imágenes binarias (número de píxeles destacados en ambas imágenes a la vez) y P es la imagen predicha.
Parámetros para evaluación de la detección y clasificación de objetos (o instance segmentation):
La instance segmentation intenta detectar objetos en una imagen y distinguirlos tanto del fondo como del resto de objetos. De nuevo, hay varios parámetros que se pueden utilizar:
- Es habitual calcular un valor de IoU entre la predicción y la ground-truth objeto a objeto. Esto permite identificar verdaderos positivos (NbTrue positive, normalmente con IoU>0,5), falsos positivos (NbFalse positive) y falsos negativos (NbFalse negative):

Donde NbGround-truth y NbPrediction son el número de objetos en la ground-truth y en la predicción, respectivamente.
- La Precisión se define como el número de objetos segmentados correctamente dividido por el número total de objetos detectados. Permite valorar el coste de los falsos positivos. Cuanto más se acerque el resultado a 1, mejor será la predicción de la red neuronal.
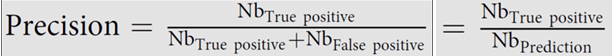
- El Recall calcula cuántos de los objetos positivos reales ha reconocido el modelo como verdaderos positivos. Se puede usar para evaluar el coste asociado a los falsos negativos. Cuanto más se acerque a 1, mejor.

- El F1 combina los valores de Precisión y Recall en un único parámetro.

- La mean matched score es la media de los IoU de los verdaderos positivos. La mean true score es el mismo valor, pero normalizado al número total de objetos en la ground-truth. La Panoptic Quality es el producto de F1 y la mean true score.
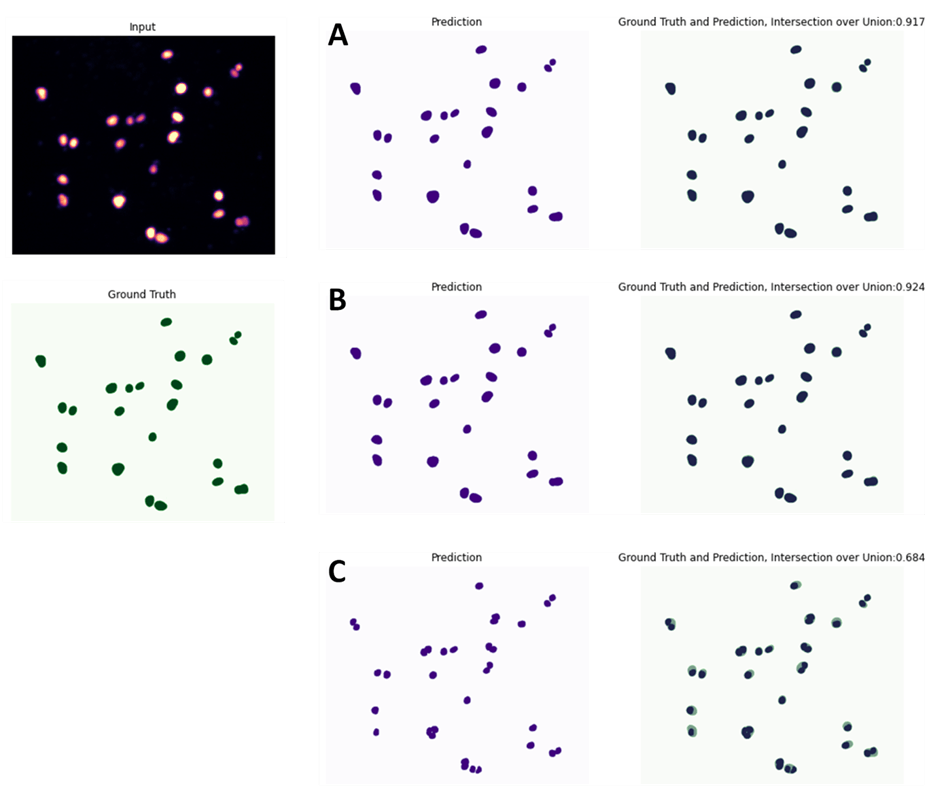
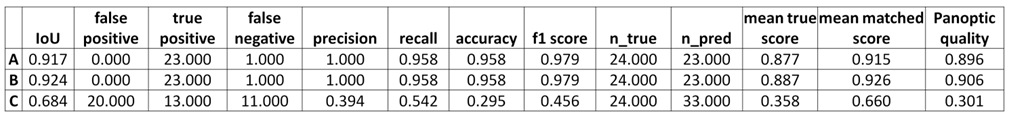
En la figura anterior vemos el resultado de aplicar a la imagen «Input» tres modelos de instance segmentation distintos entrenados con StarDist (para más detalles sobre StarDist podéis ver la newsletter(7) que publicamos anteriormente y el artículo original de Schmidt y col. 2018(8)). En la tabla podemos ver los valores de detección y segmentación para cada uno de los tres modelos. Observamos cómo el modelo C ofrece unos resultados bastante pobres tanto en la segmentación (con una IoU de 0,684), como en el reconocimiento de objetos (con gran número de falsos positivos y falsos negativos). En cambio, los modelos A y B producen unos resultados muy buenos y prácticamente idénticos, solo diferenciándose ligeramente en el valor de IoU (y, por consiguiente, en los valores que dependen de la IoU para su cálculo).
En resumen, en esta newsletter hemos visto cómo, con la ayuda de los de los Colab Notebooks del proyecto ZeroCostDL4Mic, se han evaluado distintos modelos de ANNs de una manera sencilla. Los parámetros de evaluación propuestos producen unos valores que permiten una fácil valoración de la validez de los modelos de ANNs , permitiendo la elección de los más apropiados. Además, los mapas de SSIM y RSE facilitan la detección de posibles artefactos en la reconstrucción o mejora de las imágenes.
Por último, quería resaltar que los Colab Notebooks de ZeroCostDL4Mic no solo se han usado para la evaluación, sino también para entrenar las distintas ANNs que hemos empleado hoy en los ejemplos. En una próxima newsletter os hablaré de este interesante proyecto que pone el uso de la IA para el análisis y mejora de las imágenes científicas al alcance de todo el mundo, ya que no requiere ni experiencia en programación ni un equipo informático especial y es completamente abierto y gratuito.
Referencias
- Laine RF, Arganda-Carreras I, Henriques R, Jacquemet G. Avoiding a replication crisis in deep-learning-based bioimage analysis. Nat Methods. 2021 Oct;18(10):1136-1144. doi: 10.1038/s41592-021-01284-3. PMID: 34608322; PMCID: PMC7611896.
- von Chamier L, Laine RF, Jukkala J, Spahn C, Krentzel D, Nehme E, Lerche M, Hernández-Pérez S, Mattila PK, Karinou E, Holden S, Solak AC, Krull A, Buchholz TO, Jones ML, Royer LA, Leterrier C, Shechtman Y, Jug F, Heilemann M, Jacquemet G, Henriques R. Democratising deep learning for microscopy with ZeroCostDL4Mic. Nat Commun. 2021 Apr 15;12(1):2276. doi: 10.1038/s41467-021-22518-0. PMID: 33859193; PMCID: PMC8050272
- https://github.com/HenriquesLab/ZeroCostDL4Mic/wiki
- Weigert, M., Schmidt, U., Boothe, T. et al. Content-aware image restoration: pushing the limits of fluorescence microscopy. Nat Methods 15, 1090–1097 (2018). https://doi.org/10.1038/s41592-018-0216-7
- Jaakko Lehtinen, Jacob Munkberg, Jon Hasselgren, Samuli Laine, Tero Karras, Miika Aittala, Timo Aila. Noise2Noise: Learning Image Restoration without Clean Data. https://arxiv.org/abs/1803.04189
- Mazarío J. Mejora de imágenes ruidosas mediante inteligencia artificial. https://saishnp.com/2021/04/15/mejora-de-imagenes-ruidosas-mediante-inteligencia-artificial/
- Mazarío J. StarDist: Detección de objetos en imágenes mediante deep learning https://saishnp.com/2020/10/19/stardist-deteccion-de-objetos-en-imagenes-mediante-deep-learning/
- Schmidt, U., Weigert, M., Broaddus, C., Myers, G. (2018). Cell Detection with Star-Convex Polygons. In: Frangi, A., Schnabel, J., Davatzikos, C., Alberola-López, C., Fichtinger, G. (eds) Medical Image Computing and Computer Assisted Intervention – MICCAI 2018. MICCAI 2018. Lecture Notes in Computer Science(), vol 11071. Springer, Cham. https://doi.org/10.1007/978-3-030-00934-2_30. https://arxiv.org/abs/1806.03535
AUTOR
Javier Mazarío Torrijos
Responsable del Servicio de Microscopía y Análisis de Imagen
Hospital Nacional de Parapléjicos.
Toledo, España.
Para más información puedes contactar conmigo en: jmazario@sescam.jccm.es
Si quieres conocer más sobre nosotros: https://hnparaplejicos.sanidad.castillalamancha.es/es/profesionales/investigacion/servicios-apoyo/microscopia
Síguenos en:
www.linkedin.com/in/servicios-de-apoyo-a-la-investigación-sais-hnp
Un comentario en “VALIDACIÓN DE MODELOS DE INTELIGENCIA ARTIFICIAL PARA EL ANÁLISIS DE IMÁGENES BIOMÉDICAS”