En la newsletter del 4 de octubre de 2021 os hablé del programa informático QuPath explicando su capacidad para trabajar con imágenes de gran tamaño y su sencilla integración con ImageJ. También indiqué entonces que habría una segunda parte en la que profundizaríamos más en las capacidades de este programa, que es mucho más que un simple navegador de imágenes de gran tamaño, y eso es lo que vamos a tratar en el artículo de hoy.
En primer lugar, os recuerdo que QuPath es un programa de código abierto para el análisis de bioimágenes desarrollado por Pete Bankhead (que se encuentra actualmente en la Universidad de Edimburgo). Aunque está pensado para aplicaciones de patología digital por su facilidad para trabajar con imágenes de alta resolución de portas enteros, realmente se puede utilizar para muchas más cosas.
El programa cuenta con potentes herramientas de anotación y visualización, algoritmos para la detección de tejido y células, machine learning interactivo para la clasificación de píxeles y objetos y es compatible con distintas herramientas de código abierto (como ImageJ, por ejemplo), a la vez que puede trabajar con una gran variedad de formatos de imagen.
En la anterior newsletter vimos un ejemplo de cómo utilizar QuPath para navegar fácilmente por una imagen de gran tamaño de una sección de la médula espinal de un cerdo y seleccionar una región de interés (en este caso el asta ventral) que se exportó a la versión de ImageJ integrada en el programa para hacer una detección y medición de las neuronas presentes en esa región. Ahora vamos a ver cómo hacer una detección similar en la misma muestra usando únicamente QuPath y sin necesidad de limitarnos a una región pequeña.
Para empezar, crearemos un proyecto donde añadiremos todas las imágenes que queremos analizar. Después elegiremos una de ellas y seleccionaremos un área que es la que emplearemos para poner a punto los parámetros de detección. Se podría hacer directamente en toda la muestra, pero si esta es de gran tamaño, el proceso será muy lento.
A continuación, desde Analyze>Cell detection>Cell detection (Figura 1) determinamos los parámetros que el programa va a utilizar para detectar nuestras células (motoneuronas). Aquí podemos indicar en qué canal hacer la detección (en este caso se llama «DAPI Pentacroico», aunque no es una tinción de DAPI la que estamos observando), la resolución a la que hacer la detección, el tamaño de los objetos (bajo Nucleus parameters, aunque en este caso los objetos que estamos detectando no son núcleos), la intensidad y otra serie de valores para ayudar a la detección.
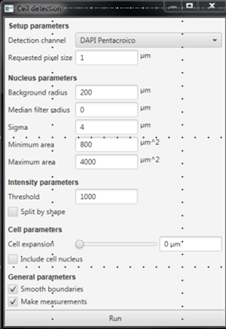
Figura 1: Ventana para poner a punto los parámetros de detección de objetos (células)
Pulsando Run vemos el resultado de esa detección en la región que habíamos seleccionado previamente (Figura 2). Una vez estemos conformes, podemos pasar a analizar una región más grande o la imagen entera (Figura 3).
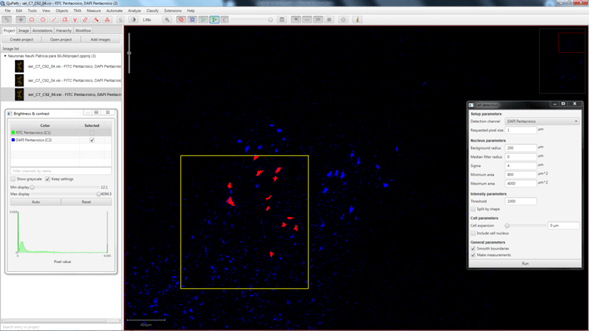
Figura 2: En rojo podemos observar los objetos detectados usando los parámetros definidos en la figura1. Los ajustes de tamaño de los objetos nos permiten excluir de la detección las neuronas demasiado pequeñas para ser motoneuronas. Muestra por cortesía de Patricia del Cerro (Grupo de Reparación Neural y Biomateriales, HNP).
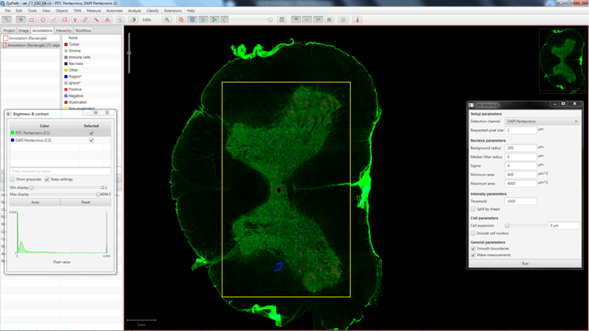
Figura 3: Resultado de aplicar la detección de objetos a una región que incluye la sustancia gris de la sección de médula espinal estudiada. Los objetos detectados se ven en rojo.
Cuando se complete el análisis podremos ir a Measure>Show detection measurements para obtener una tabla con los valores de las mediciones realizadas en cada uno de los objetos detectados (Figura 4). Estos datos se pueden guardar fácilmente para trabajar posteriormente con ellos en una hoja de cálculo.
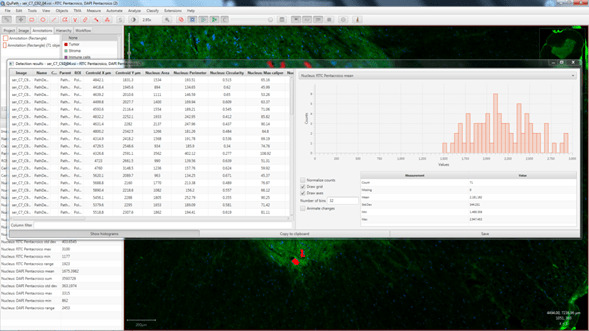
Figura 4: Tabla de resultados con las mediciones de los objetos detectados. Los datos se pueden representar en forma de histograma para los distintos parámetros. Además, se pueden exportar fácilmente para trabajar con ellos en una hoja de cálculo.
En otras ocasiones, más que detectar objetos en la muestra, nos puede interesar detectar áreas con distintos marcajes. A veces, este tipo de detección se puede hacer fácilmente utilizando un umbral de intensidad (especialmente en imágenes de fluorescencia) aunque en otras muchas ocasiones es difícil conseguir unos buenos resultados usando únicamente ese tipo de segmentación. Como os indicaba al principio del artículo, QuPath incorpora un sistema de machine learning interactivo para la clasificación de píxeles. Este sistema funciona de una manera similar al plugin de FIJI Trainable WEKA segmentation (del que ya hablamos anteriormente) y es muy fácil de entrenar.
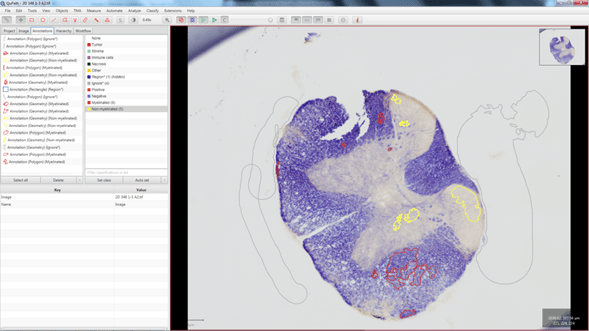
Figura 5: Imagen con una tinción de eriocomo-cianina con anotaciones Myelinated (rojo), Non myelinated (amarillo) e Ignore* (gris). Muestra por cortesía del Grupo de Neuroinmuno-Reparación, HNP
De nuevo, partiremos de un proyecto de QuPath en el que habremos metido las imágenes que queramos analizar. En nuestro ejemplo serán unas secciones de médula espinal de rata teñidas con eriocromo-cianina en las que queremos medir el área de tejido mielinizado y no mielinizado.
Empezaremos anotando algunas regiones de una de las imágenes y asignando estas regiones a las distintas categorías que queramos clasificar. En este caso empleamos las categorías Myelinated, Non-myelinated e Ignore (Figura 5). Esta tercera categoría nos permitirá detectar los píxeles de fondo y que estos no se incluyan en los cálculos de las mediciones que se hagan posteriormente.
Desde Classify>Pixel classification>Train pixel classifier accedemos a la interfaz donde podemos ajustar los parámetros para entrenar el clasificador. Pulsando el botón Live prediction podremos ver en vivo cómo está funcionando el clasificador y el efecto que tienen los cambios en los distintos parámetros. Además, podremos seguir dibujando nuevas regiones para refinar las zonas en las que el clasificador no esté detectando correctamente (Figura 6).
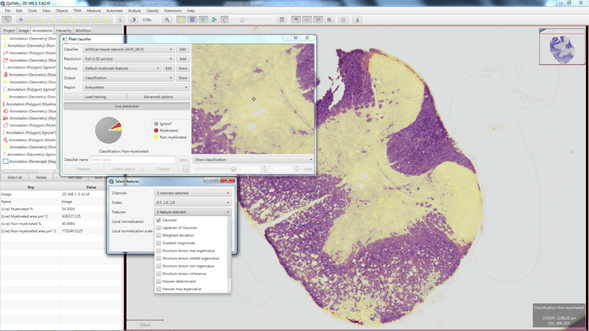
Figura 6: Predicción en vivo de la clasificación de los píxeles basada en las regiones dibujadas y los parámetros empleados para el entrenamiento.
Cuando consideremos que el clasificador está funcionando de una manera adecuada, aplicaremos la clasificación para realizar mediciones por separado en cada una de las categorías detectadas (Figura 7). Además, este clasificador se puede guardar para aplicarlo al resto de imágenes del proyecto.
En el ejemplo que estamos viendo, cada píxel de la imagen quedará asignado a una de las tres categorías que hemos definido (Myelinated, Non-myelinated e Ignore), pero, como indicamos anteriormente, los píxeles clasificados como Ignore no se tendrán en cuenta para las mediciones.
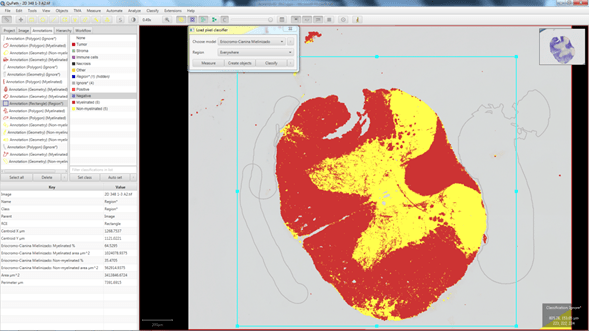
Figura 7: Resultados obtenidos al aplicar el clasificador entrenado para detectar tejido mielinizado y no mielinizado. Los píxeles clasificados como Ignore no se tienen en cuenta a la hora de realizar las mediciones. En este caso, hemos restringido la medición al área definida por el cuadrado azul.
En esta newsletter os he mostrado cómo se puede usar QuPath para detectar, de una manera bastante sencilla, tanto objetos como regiones en distintas muestras de tejido. Espero que os haya resultado interesante y que os animéis a usarlo. Para una descripción mucho más detallada (paso a paso) de cómo realizar estos procesos, os recomiendo que le echéis un vistazo a la excelente documentación del programa en el siguiente enlace:
https://qupath.readthedocs.io/en/stable/index.html
Para descargar el programa podéis ir a:
https://qupath.github.io/
Aquí un webinar donde Pete Bankhead nos habla de sus posibilidades:
https://youtu.be/4An5n6Y_rRI
Tutoriales:
https://www.youtube.com/channel/UCqepVnS1QsB7B8nBA9T91EQ/videos
Y, por último, el enlace al artículo original, para que lo citéis en vuestras publicaciones con este programa:
https://doi.org/10.1038/s41598-017-17204-5
AUTOR
Javier Mazarío Torrijos
Responsable del Servicio de Microscopía y Análisis de Imagen
Hospital Nacional de Parapléjicos.
Toledo, España.