En anteriores newsletters, os hemos ido introduciendo a las diferentes opciones de experimentos en proteómica cuantitativa y la importancia de la identificación y cuantificación integral de todas las proteínas en un sistema biológico, para revelar las funciones de las proteínas en los procesos biológicos, fisiológicos y patológicos. En esta nueva entrega nos enfocamos en los métodos basados en marcaje o proteómica cuantitativa multiplexada, con etiquetado o marcaje químico basada en métodos de adquisición de espectros de MS2.
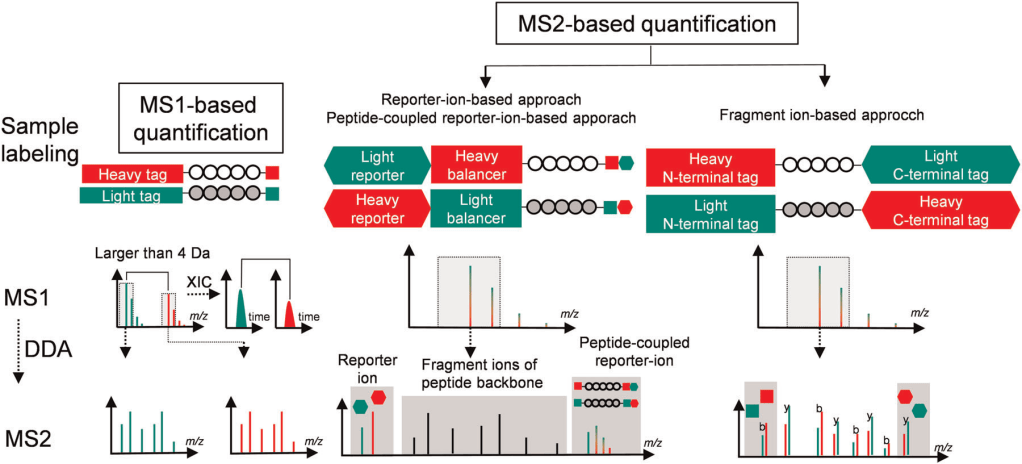
Figure 1. Descripción esquemática de la cuantificación basada en MS1 y MS2. (Xiaobo Tian et al., Mass Spectrom Rev. 2021;1–31)
CUANTIFICACIÓN BASADA EN MS2
Los enfoques de cuantificación basados en MS2 permiten fácilmente una mayor capacidad de multiplexación sin la mayoría de los inconvenientes de los métodos de cuantificación basados en MS1. La cuantificación basada en MS2 generalmente se basa en el etiquetado de los mismos péptidos derivados de diferentes muestras con diferentes etiquetas isobáricas.
Las etiquetas isobáricas son reactivos que se utilizan para modificar péptidos de forma covalente, mediante la distribución de isótopos pesados en la etiqueta para codificar diferentes condiciones, generalmente se agregan después del proceso de digestión. Consisten en grupos de iones informadores (Reporter) y de iones de equilibrado de masas.
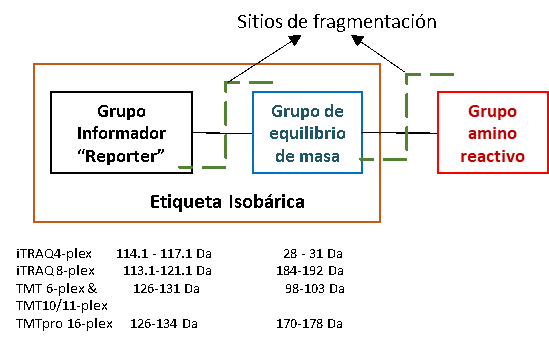
Cada variante de un conjunto de etiquetas isobáricas tiene una masa total idéntica. La única diferencia es cómo se distribuyen los isótopos pesados entre el ion “reporter” y el ion de balance de masa.
Así, un mismo péptido de diferente muestra eluye a un mismo tiempo y aparece como una única señal en el espectro MS1.
Esta es una gran ventaja porque la complejidad de los espectros de MS1 no aumenta significativamente con el número de muestras. Esto hace que las etiquetas isobáricas sean compatibles con una mayor multiplexación (actualmente hasta 16) en comparación con, por ejemplo, SILAC. Después de aislar un péptido y producir su escisión, en el espectro de MS2 observaremos los iones indicadores/informadores mostrando diferente masa, que se emplean en la cuantificación relativa; y los iones b e y que se utilizan para la identificación del péptido.
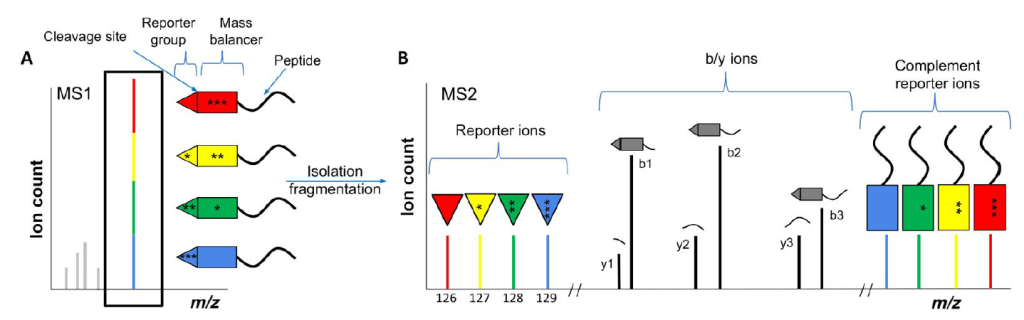
Figure 2 Esquema de proteómica multiplexada con etiquetas isobáricas. A) Las etiquetas isobáricas tienen la misma masa total, pero diferentes distribuciones de isótopos pesados entre el grupo informador y el balance de masas. Los isótopos pesados se muestran como asteriscos.
TIPOS DE ETIQUETAS
Las etiquetas isobáricas más utilizadas son la etiqueta de masa en tándem (TMT) y la etiqueta isobárica para cuantificación relativa y absoluta (iTRAQ), pero existen otras etiquetas isobáricas que han ido surgiendo para mejorar de este tipo de cuantificación.
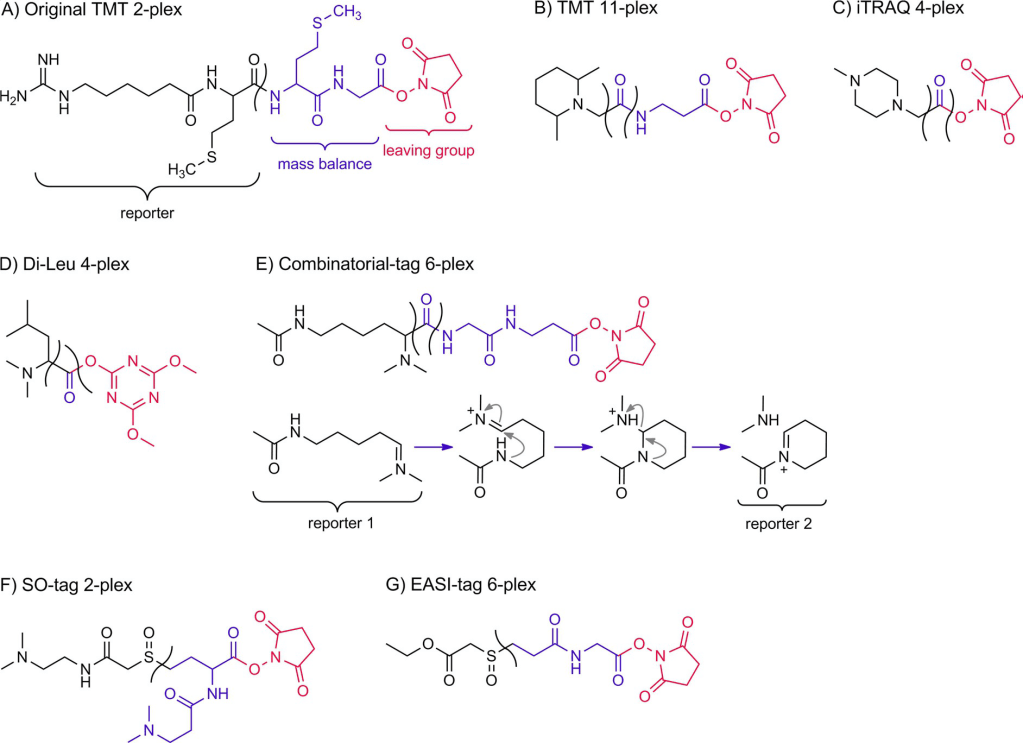
Figure 3 Descripción general de las etiquetas isobáricas. La parte negra de la estructura indica la parte informadora; el grupo de balance de masa es azul; y el grupo saliente, que se elimina después de que la etiqueta reacciona con los péptidos, es rojo.
El diseño de estas nuevas etiquetas se basa en la distribución de isótopos pesados por su estructura. De esta forma al fragmentarse, se producen iones indicadores de m/z baja, pero también el péptido intacto con el grupo de balance de masa marcado que nos aportan información complementaria y que pueden utilizarse en la cuantificación.
Lo que se busca con estos nuevos marcadores isobáricos es encontrar métodos más selectivos para introducir la etiqueta, reducir el coste de síntesis y aumentar la sensibilidad de la medida.
Uno de los problemas o desventajas de la proteómica multiplexada es la distorsión de la relación de medida. Incluso para la ventana de aislamiento técnicamente más pequeña posible centrada en un péptido de interés, en un experimento real, se coaislarán otros péptidos con m/z y tiempo de retención similares. Por tanto, casi todas las mediciones están distorsionadas, aunque tiende a ser más significativa para los péptidos de baja abundancia.
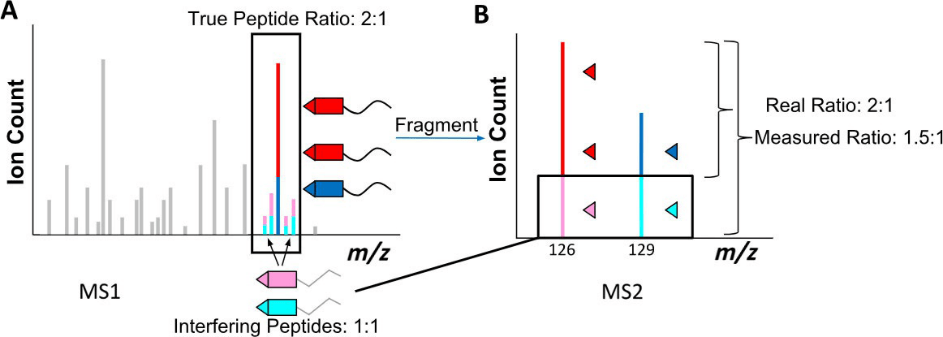
Figure 4 El problema de la proteómica multiplexada: distorsión de la relación
El problema de la distorsión puede aliviarse con enfoques aplicables durante la preparación de la muestra (mayor fraccionamiento de la muestra por LC), el análisis de MS (fragmentación MS3) o el procesamiento computacional una vez adquiridos los datos.
PROTEÓMICA CUANTITATIVA CON MARCAJE ISOBÁRICO EN LESIÓN MEDULAR
Uno de los estudios más recientes en la literatura que implica lesión medular y marcaje isobárico es el de Hulme, C. H., et al Spinal Cord, 1-6 – October 2021 en el que caracterizan como cambia el proteoma del plasma después de una LME completa en comparación con una lesión simulada en modelos de rata. En el que identifican como vías reguladas diferencialmente, la señalización de la respuesta de fase aguda desencadena por citoquinas inflamatorias y cambios en la activación de LXR/RXR destacando la importancia de considerar el efecto de la LME en otros órganos como el hígado.
Otro estudio, es el Liu S et al Med Sci Monit 2020; 26:e924266, en el que examinan la expresión de proteínas en un modelo de LME por contusión después de una semana de provocar la lesión. Para ello realizan un marcaje isobárico con iTRAQ y posterior análisis por LC-MS/MS. Algunas de las proteínas diferenciadas en el estudio ya las habían detectado en un estudio anterior (Zhou H., et al 2018) en el que utilizaban iTRAQ para examinar los niveles de expresión de proteínas 14 días después de la LME describiendo el perfil proteómico de la fase de transición entre la fase subaguda a la fase intermedia de la LME.
Tabla 1 Las 10 mejores proteínas sobreexpresadas y disminuidas

Quizá, algunas de estas proteínas puedan ser potenciales biomarcadores para el diagnóstico y tratamiento una semana después de LME.
CONCLUSIÓN
En comparación con otros enfoques de etiquetado de isótopos estables, la tecnología de etiquetado isobárico tiene muchas ventajas. En primer lugar, el etiquetado isobárico tiene una mayor capacidad de multiplexación (hasta 16), lo que aumenta considerablemente el rendimiento de la cuantificación. En segundo lugar, la alta capacidad de multiplexación del marcaje isobárico hace posible manejar varias réplicas biológicas y ofrecer datos de validación estadística con un experimento de cromatografía líquida (LC)-MS/MS.
Los enfoques de marcado isobárico son compatibles con casi todos los sistemas biológicos, incluidas las células, los tejidos y los biofluidos.
Aunque la flexibilidad y las capacidades de multiplexación hacen que los métodos de marcaje isobárico sean particularmente adecuados para aplicaciones biológicas, sufren de precisión y exactitud reducidas. Los términos «precisión» y «exactitud» se refieren a la reproducibilidad de la medición y la proximidad al valor real de un cambio, respectivamente.
AUTOR
Gemma Barroso García, MSc.
Responsble SAI-Proteómica.
Hospital Nacional de Parapléjicos.
Toledo, España.
Referencias
- Pappireddi N., Martin L., Wühr M. A Review on Quantitative Multiplexed Proteomics. ChemBioChem 2019, 20, 1210-1224 https://doi.org/10.1002/cbic.201800650
- Tian X, Permentier HP, Bischoff R. Chemical isotope labeling for quantitative proteomics. Mass Spectrom Rev. (2021);1‐31. https://doi.org/10.1002/mas.21709