Recientemente hemos adquirido en el SMAI un microscopio Olympus que se controla a través del software CellSens Dimensions. El programa nos permite capturar mosaicos de imágenes de grandes áreas a alta resolución espacial y con una profundidad de hasta 16-bit por canal. Esto nos ha abierto grandes posibilidades en cuanto a la capacidad de adquisición de imágenes pero también presenta nuevos retos ya que el tamaño resultante de estos archivos es masivo (varios GB por imagen).
Por defecto, las imágenes se guardan con el formato de Olympus «.vsi» (acrónimo de virtual slide image). Este formato es excelente para trabajar con imágenes de gran tamaño ya que almacena la información de un modo piramidal, es decir, la imagen se guarda con varias capas, cada una de ellas con menor resolución que la anterior de modo que no es necesario para el ordenador cargar la imagen completa en todo momento sino que se va adaptando según se va navegando por la imagen.

Figura 1: Ejemplo de la estructura de una imagen piramidal. Imagen obtenida de: https://en.wikipedia.org/wiki/File:Image_pyramid.svg
El análisis de las imágenes se puede realizar con el mismo programa que se usa para la captura (CellSens Dimensions). El inconveniente que presenta esta forma de trabajo es que es necesario tener licencias individuales que habiliten la parte de análisis de imagen del software, y estas licencias no son fácilmente trasladables entre equipos, aparte de tener un coste económico elevado.
Desde hace tiempo, el programa de análisis de imagen de referencia en nuestro centro es ImageJ/FIJI, tanto por su gran versatilidad como por el hecho de que es gratuito. Gracias al plugin «BioFormats», FIJI puede convertir fácilmente los archivos «.vsi» en archivos «.tif» que posteriormente se pueden analizar. El problema que surge es que el formato tif tiene varias limitaciones. La primera es que el tamaño máximo que pueden tener es de 2GB por capa por lo que si la imagen original tiene un tamaño mayor que ese, no se podrá convertir a formato tif. Además, el trabajo con estas imágenes tan grandes es poco fluido ya que operaciones sencillas como un cambio de canal o ajustes de brillo/contraste o de selección de umbrales pueden llevar decenas de segundos incluso cuando se emplean ordenadores potentes, lo que dificulta enormemente la capacidad de trabajar de un modo eficiente con ellas.
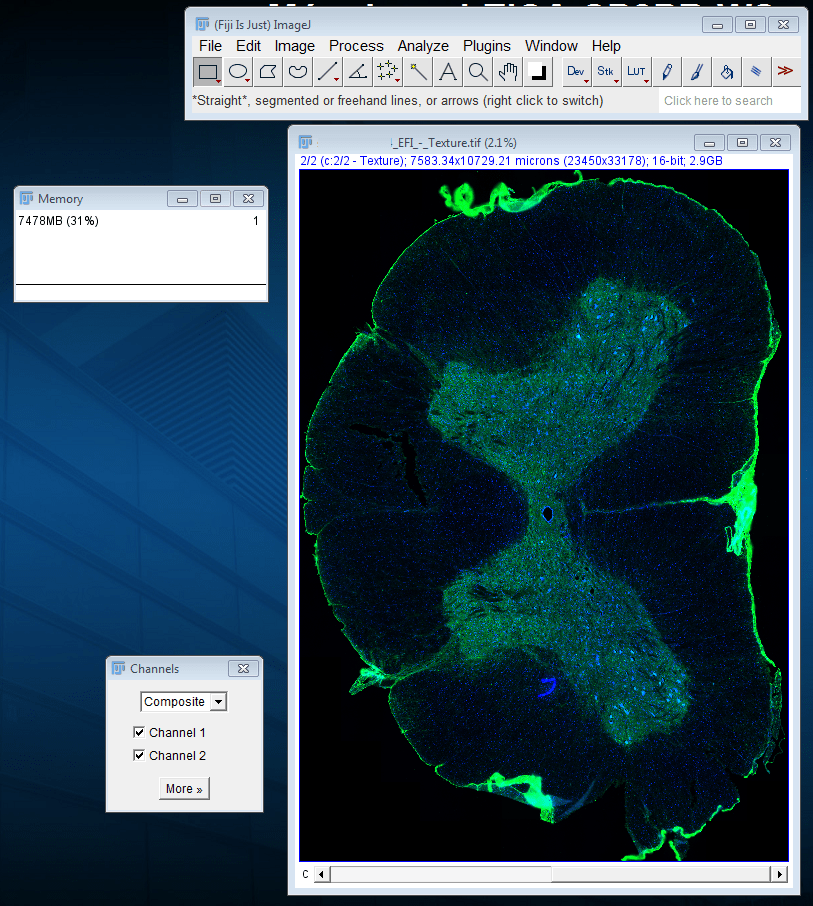
Figura 2: Esta imagen de dos canales pesa 2,9 GB (1,45 GB por canal) pero requiere el uso de casi 7,5GB de memoria RAM. El manejo de estas imágenes es poco fluido, lo que hace que sea complicado trabajar con ellas. Foto por cortesía de Patricia del Cerro (Grupo de Reparación Neural y Biomateriales, HNP).
Para poder visualizar de una manera práctica conjuntos de datos de gran tamaño (hasta del orden de TB) como los que se obtienen con los escaneadores de portas o los microscopios de light sheet, se está desarrollando dentro de FIJI un ecosistema de plugins, conocido como Big Data Viewer (BDV), en el que los datos se organizan con una resolución piramidal (como veíamos en la figura 1), lo que facilita la navegación por las imágenes, aunque la capacidad de procesado y análisis es todavía limitada dentro del ecosistema si la comparamos con la de FIJI (pero es algo que está en desarrollo y que mejorará con el tiempo), así que sigue sin darnos la herramienta que nosotros necesitamos.
Afortunadamente, existe un programa que, al igual que FIJI, es libre y que permite trabajar a la perfección con el tipo de imágenes que nos interesan. Se llama QuPath y está pensado para trabajar fácilmente con imágenes de alta resolución de portas enteros.
Siempre da un poco de vértigo enfrentarse a un nuevo programa, especialmente cuando (como es mi caso) se lleva años aprendiendo a manejar y usando otro para hacer el análisis de las imágenes, pero hay que resaltar que el autor de QuPath, Pete Bankhead (que se encuentra actualmente trabajando en el Departamento de Patología de la Universidad de Edimburgo), se confiesa un admirador de ImageJ (de hecho, el proyecto comenzó como una serie de plugins para ImageJ aunque luego evolucionó a un nuevo programa) y ha incorporado en QuPath muchos de los atributos y formas de trabajo que considera más útiles de ImageJ.
Un ejemplo de esto es que para abrir una imagen basta con arrastrarla sobre el programa para que este la abra automáticamente. Esta imagen conserva su formato piramidal, lo que hace que la navegación sobre la misma sea muy fluida incluso en equipos sin mucha potencia o cuando las imágenes están ubicadas en un servidor o en equipos conectados en red. Además, las regiones de interés que se seleccionen en QuPath se pueden exportar fácilmente a una versión de ImageJ que está incluida en el programa (Figura 3).

Figura 3: La región de interés (rectángulo amarillo) se puede exportar a ImageJ pulsando el botón señalado con la flecha amarilla. A continuación se puede decidir si se exporta con resolución completa o si se aplica un factor de reducción de resolución.
De este modo, operaciones que se sepan hacer en ImageJ, o incluso macros que se tengan ya escritas para este programa, se pueden ejecutar sin necesidad de aprender a hacerlas de nuevo en QuPath (Figura 4). Esta es una característica que facilita enormemente la toma de contacto con QuPath para los usuarios con experiencia con ImageJ/FIJI.
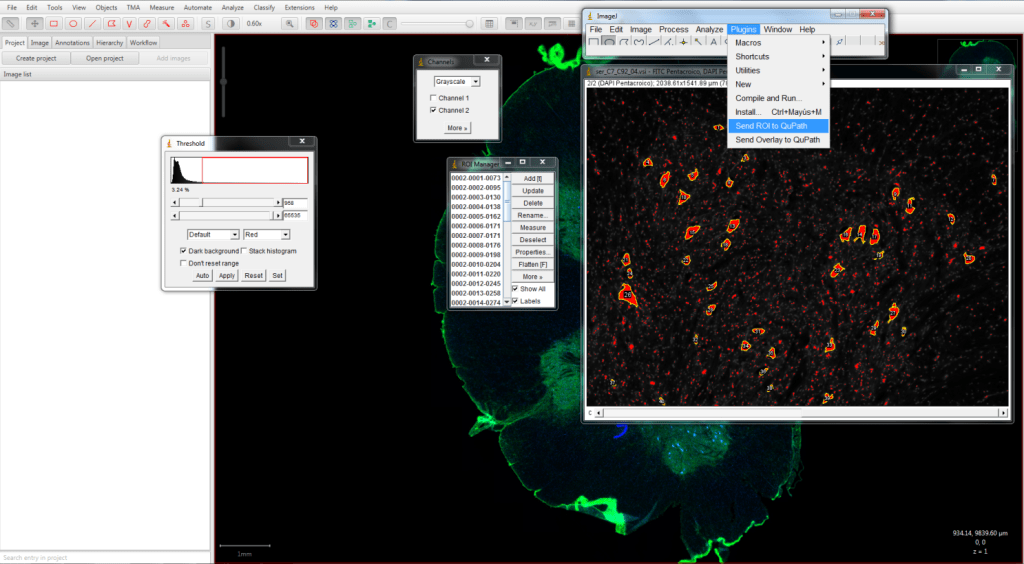
Figura 4: Análisis de partículas realizado sobre el canal 2 de la región exportada de QuPath a ImageJ. Los resultados se pueden exportar de nuevo a QuPath (flecha amarilla).
La comunicación entre QuPath e ImageJ funciona en los dos sentidos, de modo que los resultados obtenidos de esta manera se pueden enviar de nuevo a QuPath para continuar el análisis con este programa (Figuras 4 y 5).
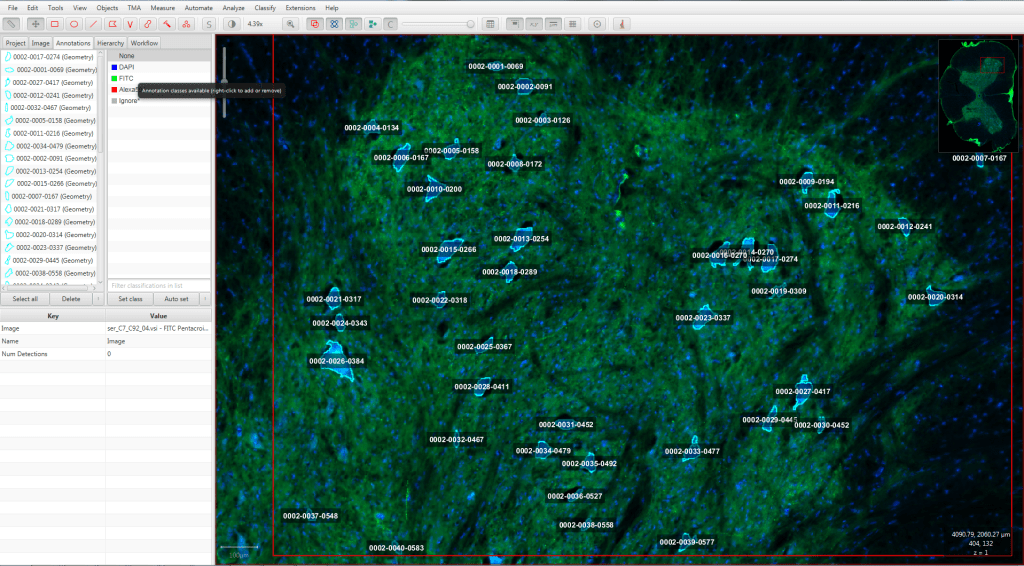
Figura 5: Detalle de las células detectadas con ImageJ e importadas como anotaciones a QuPath.
Pero QuPath es mucho más que un navegador de imágenes de gran tamaño con integración con ImageJ. Es una potente herramienta de anotación de imágenes y contiene algoritmos propios para detección de tejido y células. También permite aplicar machine learning para la clasificación de píxeles (de una manera relativamente similar a la Trainable Weka Segmentation que explicamos hace tiempo) y objetos. Incluso permite el uso de modelos de deep learning desarrollados con StarDist para la detección de núcleos (también os hemos hablado de StarDist anteriormente).
En estos momentos estoy en proceso de aprendizaje del manejo del programa y en un futuro escribiré otra entrada explicando más en detalle cómo trabajar con él, pero quería que supierais de su existencia porque, si os habéis enfrentado al problema de trabajar con imágenes de gran tamaño, esta es una excelente herramienta. Y su capacidad de intercomunicación con ImageJ hace que comenzar a utilizarla para usuarios con experiencia en este último programa sea relativamente sencillo (algunos de nuestros usuarios ya están usando QuPath y están encantados). Además, espero que esto también sirva para que a algunos de vosotros os pique el gusanillo y decidáis darle una oportunidad.
Existe una excelente documentación online donde se explica claramente todo lo que hace falta saber para manejar este programa. Si estáis interesados en usarlo, os recomiendo que le echéis un vistazo:
https://qupath.readthedocs.io/en/latest/
En el siguiente enlace podéis descargar el programa:
Aquí un webinar donde Pete Bankhead nos habla de sus posibilidades:
Tutoriales:
https://www.youtube.com/channel/UCqepVnS1QsB7B8nBA9T91EQ/videos
Y, por último, el enlace al artículo original, para que lo citéis en vuestras publicaciones con este programa:
https://doi.org/10.1038/s41598-017-17204-5
AUTOR
Javier Mazarío Torrijos
Responsable del Servicio de Microscopía y Análisis de Imagen
Hospital Nacional de Parapléjicos.
Toledo, España.
Un comentario en “¿CÓMO TRABAJAR CON IMÁGENES CIENTÍFICAS DE GRAN TAMAÑO? QUPATH PUEDE AYUDARNOS”