La cuantificación de la producción de RNA mensajero (mRNA) es clave para comprender las respuestas inmediatas de las células a cambios en el ambiente, o en respuesta a un tratamiento. La respuesta celular a diferentes estímulos puede ser muy heterogénea en diferentes células o incluso dentro de la misma población.
Tradicionalmente, la expresión génica se cuantifica analizando el mRNA correspondiente mediante técnicas de RT-PCR cuantitativa, Northern Blot o Hibridación in situ con fluorescencia (FISH), o más recientemente por RNAseq, todas ellas técnicas de punto final que en la mayor parte de los casos requieren la lisis de las células, o al menos la fijación y permeabilización de las mismas.
En el caso de la RT-PCR, vamos a cuantificar la cantidad media de mRNA dentro de una muestra completa, independientemente de las poblaciones y subpoblaciones celulares que la compongan, de forma que si una subpoblación pequeña de células responde a un tratamiento concreto incrementando la expresión de un determinado gen, este cambio puede verse enmascarado por la aportación al RNA total de otras subpoblaciones que pueden ser más abundantes en la muestra y que no están respondiendo al tratamiento, obteniendo, por tanto, datos sesgados.
Tal y como vimos el año pasado en la Newsletter de Citometría Genómica, podemos analizar la expresión de RNA a nivel de célula única separando por FACS células únicas de las poblaciones de interés, y analizando su transcriptoma por secuenciación masiva (RNAseq). En esta Newsletter vamos a explicaros una aproximación diferente para analizar la expresión de determinados RNAs de interés en combinación con marcaje de proteínas, a nivel de célula única por citometría de flujo
FLOW FISH-RNA FLOW
La técnica de Flow-FISH o RNA Flow se basa en la utilización de sondas de oligonucleótidos específicos de una secuencia concreta de RNA (ya sea mRNA, miRNA, RNA viral, siRNAs, etc), que van a hibridar en el interior de la célula con sus RNA específicos. La determinación de la presencia del RNA de interés junto con la detección simultánea de proteínas en las células se realiza posteriormente por citometría de flujo, permitiendo incluso la separación de poblaciones celulares específicas que contienen el RNA de interés, para su utilización en experimentos posteriores. Además, nos va a permitir correlacionar la expresión de proteína y de RNA a nivel de célula única.
Actualmente existen dos tipos de ensayos que se diferencian principalmente en la utilización de células fijadas y permeabilizadas o de células vivas: Prime-Flow (ThermoFisher)/RNAscope (ACD) y SmartFlare (Millipore).
FLOW_FISH Prime-Flow/RNAscope
Esta técnica está basada en la técnica de hibridación in situ normal, con la diferencia de que se utiliza un sistema de amplificación de la señal fluorescente. En la realización de un FISH normal se utilizan altas temperaturas de anillamiento que provocan un aumento de la autofluorescencia, y los parámetros de dispersión de luz (FSC y SSC) se ven comprometidos. Además, al realizar marcaje de proteínas de interés con anticuerpos marcados fluorescentemente, la señal de estos puede dañarse con la temperatura de anillamiento utilizada.
Para mejorar todos los problemas relacionados con la Tª y con la baja intensidad de la señal, se desarrolló la técnica de “branched DNA” que va a permitir detectar incluso un pequeño nº de copias de un determinado RNA, utilizando una Tº de anillamiento baja (40ºC), lo que facilita la conservación de los fluorocromos acoplados a anticuerpos para detección simultánea de proteínas.
Esta técnica utiliza una batería de sondas que son diseñadas específicamente contra el RNA de interés y que tienen una forma de Z (Fig.1). Se utilizan pares de sondas complementarias a secuencias muy próximas en el RNA, que poseen una parte libre que contiene una secuencia complementaria a la secuencia de una molécula denominada pre-amplificadora, que va a ser el “tronco” de todo el “árbol” de amplificación de la señal.
Cada una de las moléculas pre-amplificadoras puede hibridar a su vez con hasta 20 secuencias amplificadoras a las que se van a unir sondas específicas marcadas fluorescentemente, de forma que podría amplificarse la señal entre 8.000 y 16.000 veces.

Fig. 1 DNA Branched Technique (Teong Soh K and Wallace P 2018)
Este tipo de aproximaciones permite monitorizar por ejemplo, la cinética transcripcional y traduccional de manera simultánea y a nivel de célula única (Fig.2).
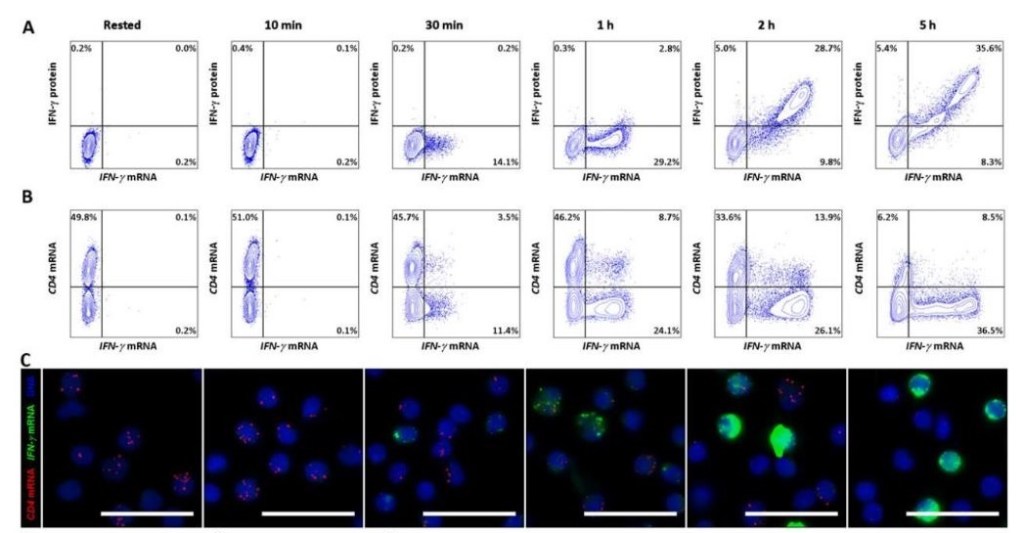
Fig. 2 Detección simultánea de 2 mRNA diferentes y la expresión de proteína intracelular: PBMCs estimuladas a diferentes tiempos con PMA+ Ionomicina fueron teñidas con sondas específicas para mRNA de CD4 y de IFNg, junto con anticuerpos específicos de IFNg (proteína intracelular) y de CD14 (marcador de monocitos, no en la figura). La co-expresión del mRNA de CD4 e IFNg se corroboró por microscopía. (Van Hoof, D et al 2014)
En la actualidad con los sistemas disponibles, pueden detectarse hasta 4 transcritos diferentes junto con todos los epítopos proteicos detectables según la configuración del citómetro en que se analicen las muestras (en nuestros citómetros podríamos detectar hasta 5 epítopos más), de forma que en una muestra heterogénea podríamos analizar, por ejemplo, la expresión de 4 transcritos diferentes en hasta 5 poblaciones distintas.
Sin embargo, como esta aproximación también es de punto final, no podríamos hacer más investigaciones con las células que están expresando nuestro RNA de interés. Para eso, existe la siguiente técnica en el mercado.
SmartFlare
Para la realización de la técnica anterior se hace necesaria la fijación y permeabilización de las células a analizar. Sin embargo, podemos estar interesados en aislar una subpoblación específica que esté expresando un RNA de nuestro interés, para poder estudiarla en experimentos y análisis posteriores.
Esto implica, por tanto, la detección de RNA en células vivas. Y para ello se ha desarrollado esta técnica SmartFlare.
Esta aproximación experimental implica la utilización de unas partículas de oro que llevan unidos varios oligonucleótidos específicos que van a hibridar con el RNA de interés que se quiere analizar (Fig.3). A estas secuencias están unidas unas sondas reporteras que cuando se encuentran unidas a ellas no son fluorescentes, y que cuando el RNA diana está presente son desplazadas por él, liberándolas al interior de la célula y activándose la fluorescencia, con lo que las células que estén expresando nuestro RNA de interés serán fluorescentes y podremos detectarlas en base a esa fluorescencia.
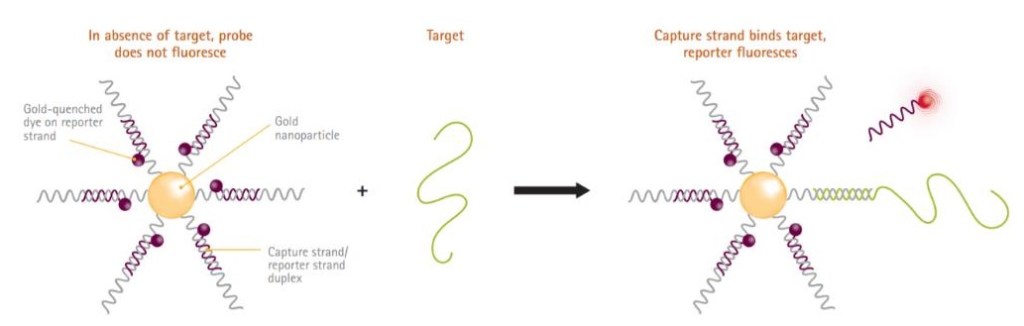
Fig. 3. Estructura de las partículas de oro y su unión al RNA diana.
Estas nanopartículas se añaden al medio de cultivo y son tomadas por las células por endocitosis activa. Esto implica que no todas las células van a ser capaces de incorporarlas, con lo que hay que hacer pruebas previamente, para asegurarse de que están incorporándolas al citoplasma. (Fig.4)
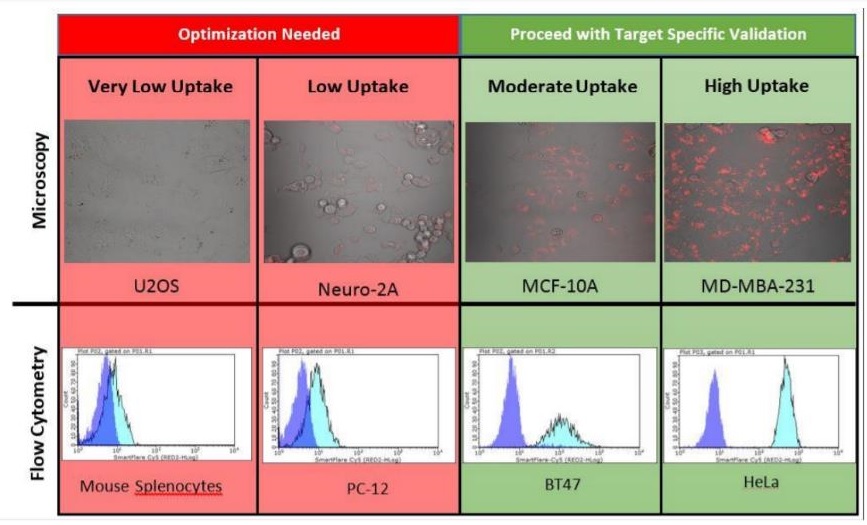
Fig.4. Ejemplo de señales obtenidas con una baja incorporación y una incorporación optimizada
Una vez marcadas las células con las nanopartículas, se puede proceder a realizar la tinción para los epítopos de interés y a analizar la muestra por citometría de flujo. Además, como las células están vivas, pueden separarse por FACS y ser utilizadas en ensayos posteriores (Fig.5).
Esta técnica permite la detección de 2 RNA diferentes (marcados con Cy3 y Cy5), combinándose con la utilización de anticuerpos específicos o sondas como el Mitotracker, sondas para detección de especies reactivas de oxígeno (ROS), etc.
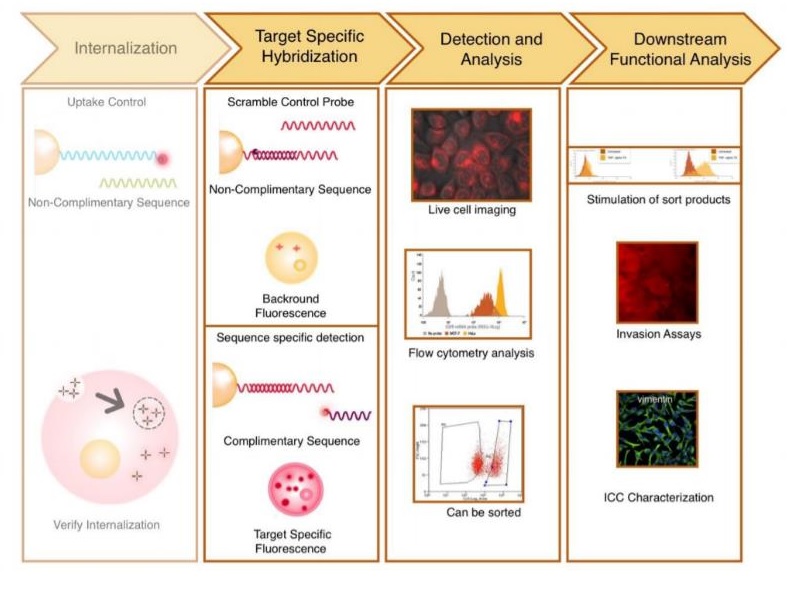
Fig. 5. Esquema del proceso completo
CONTROLES NECESARIOS
Como en toda aproximación experimental existen una serie de controles necesarios para validar la técnica. Algunos de esos controles son:
- Medición de RNAs “housekeeping”. Se deben utilizar sondas específicas y comprobar que estamos detectándolas con nuestros métodos
- Controles negativos o “scramble”. Sondas sin sentido que no se unen a ningún RNA
- Control positivo o de “uptake”. Sólo para experimentos con SmartFlare uso de nanopartículas de oro con sonda fluorescente acoplada
Y por supuesto, todos los controles habituales en cualquier marcaje multiparamétrico: controles de compensación y FMO (“Fluorescence Minus One”).
CONCLUSIÓN
La técnicas presentadas en esta newsletter van a permitir examinar el contenido de RNAs específicos a nivel de célula única, así como identificar las distintas poblaciones presentes en una muestra heterogénea que están respondiendo a un tratamiento, lesión, etc, e incluso permitir la separación y enriquecimiento de estas poblaciones celulares de interés, para posteriores ensayos o aproximaciones experimentales.
Sin embargo, estos métodos aún presentan algunas desventajas, como el tiempo invertido en el procesamiento de las muestras y el control absoluto de la Tª en el caso del Prime-Flow, o que no todas las células van a incorporar las nanopartículas en los experimentos de SmartFlare y, sobre todo, el elevado coste de reactivos.
Aun así, estas técnicas son muy sensibles, ya que permiten detectar muy pocas copias de RNA, se pueden utilizar para analizar expresión de un gen cuando no haya anticuerpos disponibles contra la proteína producida e incluso, permite utilizarlas como método de detección de partículas virales.
Y para acabar, mencionar que existen otras posibilidades de analizar ácidos nucleicos por citometría de flujo como la determinación de cariotipos y separación por sorting de cromosomas concretos, la detección del acortamiento de telómeros por FISH o incluso la detección de miRNAs específicos mediante balizas moleculares (“molecular beacons”).
AUTOR
Virginia Vila del Sol, PhD
Responsable del Servicio de Citometría de Flujo.
Hospital Nacional de Parapléjicos.
Toledo, España.
Referencias
- Soh K.T., Wallace P.K. (2018) RNA Flow Cytometry Using the Branched DNA Technique. In: Hawley T., Hawley R. (eds) Flow Cytometry Protocols. Methods in Molecular Biology, vol 1678. Humana Press, New York, NY. https://doi.org/10.1007/978-1-4939-7346-0_4
- Van Hoof, D., Lomas, W., Hanley, M.B. and Park, E. (2014), Simultaneous flow cytometric analysis of IFN-γ and CD4 mRNA and protein expression kinetics in human peripheral blood mononuclear cells during activation. Cytometry, 85: 894-900. https://doi.org/10.1002/cyto.a.22521