El high content analysis (HCA) es una técnica instrumental que emplea microscopía automatizada en combinación con análisis de imagen en un formato de alto rendimiento. En nuestro centro, esta técnica se emplea principalmente para el estudio cuantitativo de eventos celulares complejos. Como tal, uno de los primeros pasos en el análisis es identificar correctamente cada una de las células en las imágenes capturadas. Para ello tenemos que ser capaces de identificar algún tipo de estructura que tengan todas las células y que solo esté presente una vez en cada una de ellas. Habitualmente, la estructura que cumple este requisito es el núcleo celular (aunque existen, no solemos trabajar con células multinucleadas).
La identificación de los núcleos normalmente implica realizar un preprocesado de la imagen para reducir el fondo (Figura 1) seguido de una segmentación por intensidad y un postprocesado (de tipo watershed) para separar núcleos adyacentes incorrectamente identificados como uno solo (Figura 2).

Figura 1: Imagen original (izquierda) y resultado de aplicar una substracción del fondo (derecha).
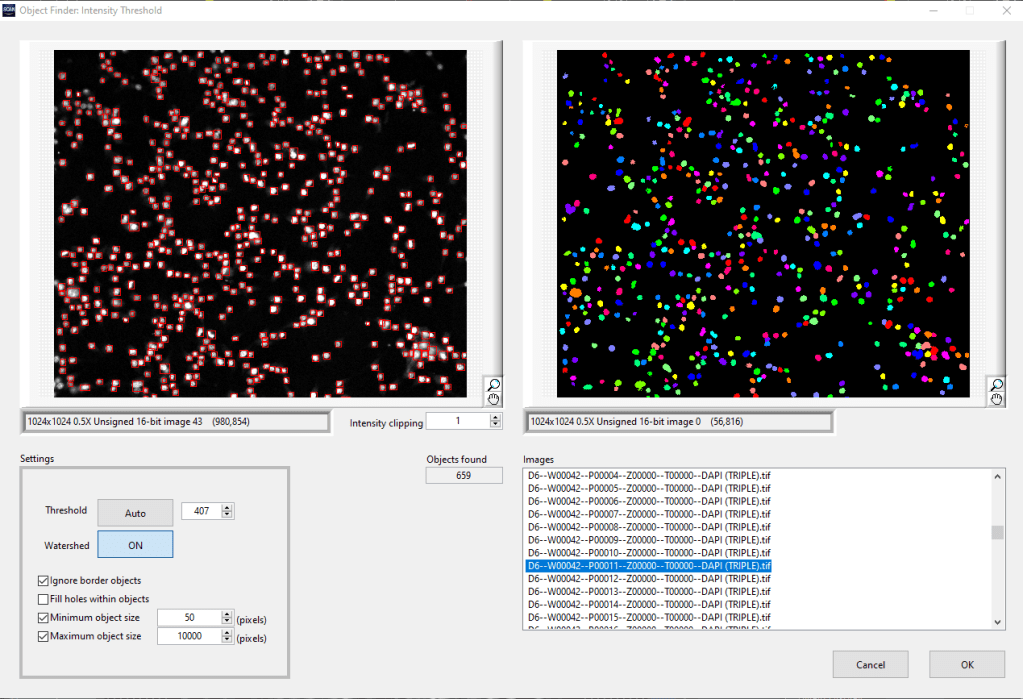
Figura 2: Resultado de la segmentación de la imagen preprocesada. Objetos detectados (izquierda) y máscara de los objetos detectados (derecha).
Debido a la variabilidad de las muestras, puede ocurrir que, si se emplean umbrales de intensidad bajos para poder detectar correctamente los núcleos con menor intensidad, no se puedan separar correctamente los núcleos adyacentes, especialmente en zonas de alta densidad celular mientras que si se emplean valores de umbral más altos que permitan separar bien los núcleos próximos, podemos perder aquellos que sean menos intensos (Figura 3).
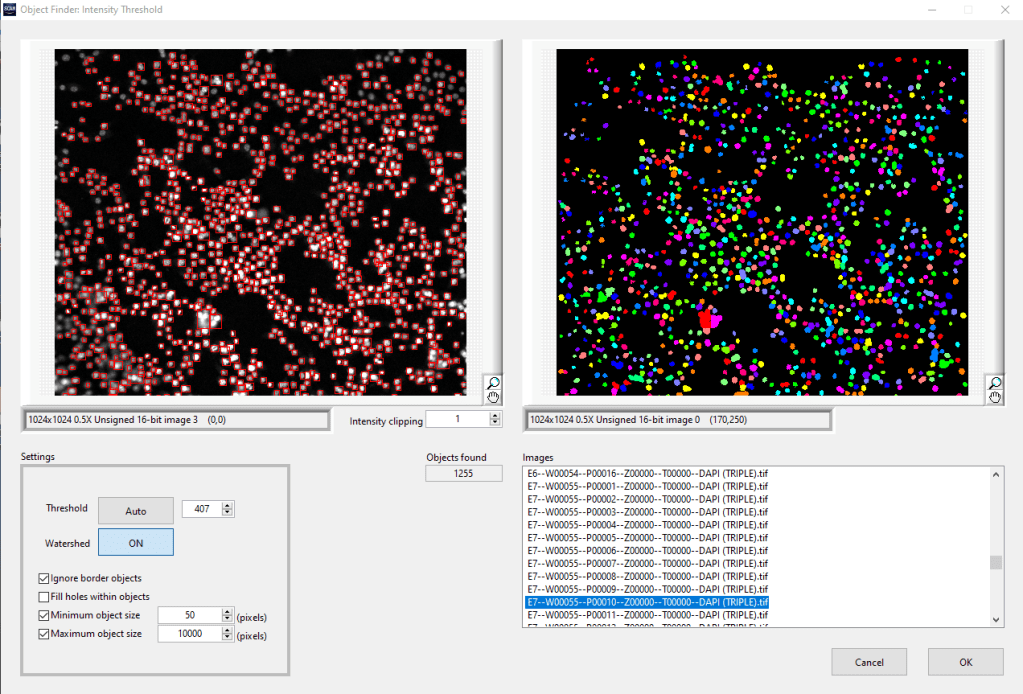
Figura 3: Los parámetros para la segmentación usados en la figura 2 no producen una buena segmentación en esta otra imagen ya que no detecta los núcleos con menor intensidad y no separa correctamente núcleos que están muy próximos.
Este problema se puede reducir gracias al módulo de análisis de imagen por inteligencia artificial con el que cuenta el programa informático ScanR de nuestro nuevo equipo Olympus para HCA. Con él, podemos entrenar una red neuronal convolucional que aprenda a identificar los núcleos.
Para realizar el entrenamiento necesitamos partir de una serie de imágenes de los núcleos junto con su solución en forma de imágenes etiquetadas, que serían lo que se conoce como ground truth. Aunque, en general, para este tipo de entrenamiento es necesario que la ground truth sea lo más precisa posible, en este caso no es tan importante que sea perfecta. De este modo, podemos utilizar como ground truth el resultado de la segmentación descrita anteriormente. Esto se debe a que lo primero que hace el programa antes de empezar a entrenar es una normalización de las imágenes. Así, los modelos resultantes son insensibles a la intensidad total de la fluorescencia. Durante las primeras iteraciones del entrenamiento, la red neuronal solo tiene tiempo de fijarse en los aspectos más característicos de la ground truth por lo que hará una buena predicción de lo que es fondo y de lo que es señal. Según vayan avanzando las iteraciones, la red neuronal tratará de predecir como fondo todos aquellos núcleos que no están anotados en la ground truth (Figura 4).
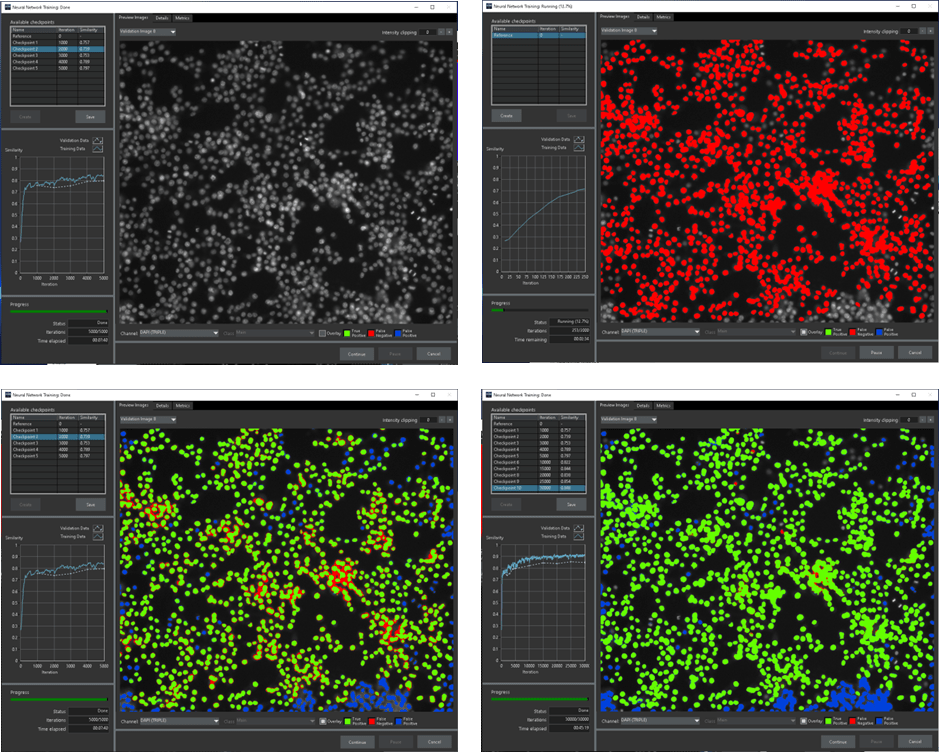
Figura 4: Una de las imágenes de validación de la red neuronal (superior izquierda) con su ground truth superpuesta (superior derecha). Con solo 2000 iteraciones de entrenamiento (inferior izquierda) la red ha aprendido a reconocer núcleos no marcados en la ground truth (azul) a la vez que separa mejor núcleos que no se segmentaron correctamente. Tras 30.000 iteraciones (inferior derecha), vemos cómo la capacidad de la red para detectar núcleos no marcados ha empeorado y, a la vez, separa peor los núcleos que teníamos problemas para separar originalmente.
El resultado del procesamiento de las imágenes con el modelo de red neuronal que hemos entrenado es una imagen en la que la intensidad de cada píxel vendrá determinada por la probabilidad de que ese píxel pertenezca a un núcleo. Esto nos proporciona imágenes con un contraste muy alto donde la segmentación es mucho más sencilla y precisa que en las imágenes originales, permitiendo una mejor identificación de los núcleos (Figura 5).
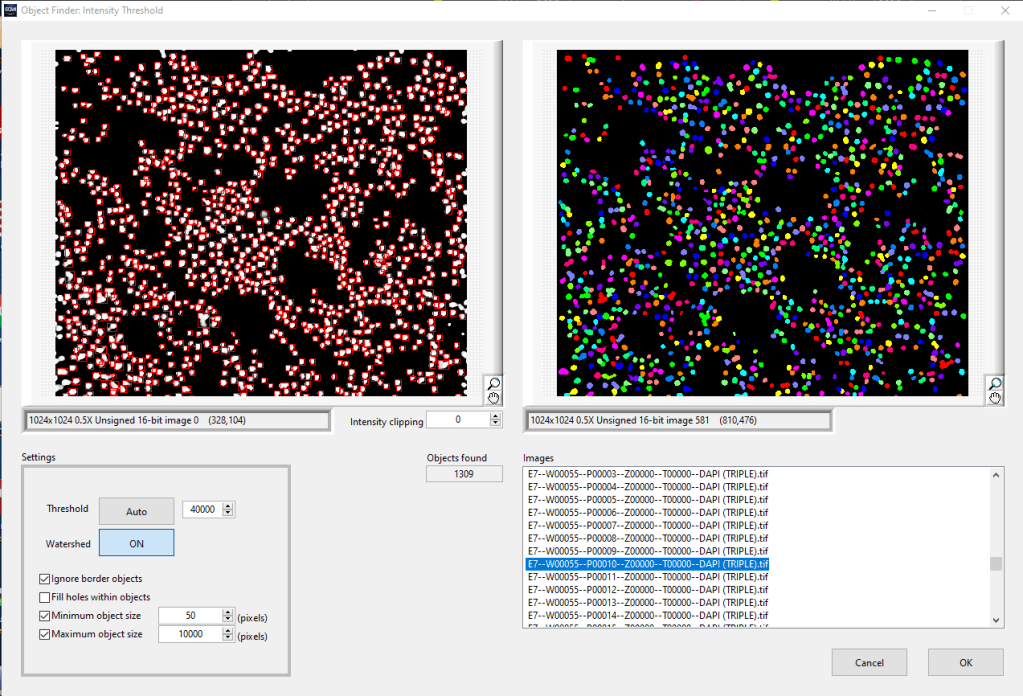
Figura 5: Segmentando sobre la imagen de probabilidad obtenida tras el procesamiento con la red neuronal convolucional que hemos entrenado, podemos hacer una mejor identificación de los núcleos, tanto los de menor intensidad como los que están muy próximos entre sí. Comparar con la figura 3.
Con los núcleos correctamente identificados podemos continuar con el análisis de nuestras muestras para poder responder a las cuestiones que plantea nuestro estudio.
En el artículo de hoy os hemos enseñado uno de los posibles usos de los modelos de inteligencia artificial que incluye ScanR, pero se pueden usar para muchas más cosas. En el siguiente enlace podéis ver un informe técnicode Olympus en el que explican cómo usar la inteligencia artificial para identificar núcleos en condiciones de muy baja iluminación:
https://www.olympus-lifescience.com/es/resources/white-papers/ultra-low_light_exposure_analysis/
Y en este otro enlace nos demuestran que se puede entrenar una red neuronal para reconocer la posición del núcleo celular en muestras sin marcaje:
https://www.olympus-lifescience.com/es/resources/white-papers/label-free_segmentation/
AUTOR
Javier Mazarío Torrijos
Responsable del Servicio de Microscopía y Análisis de Imagen
Hospital Nacional de Parapléjicos.
Toledo, España.
Un comentario en “SEGMENTACIÓN DE NÚCLEOS MEDIANTE INTELIGENCIA ARTIFICIAL EN LA PLATAFORMA DE HIGH CONTENT ANALYSIS SCANR”