Durante la última década el campo de la citometría de flujo ha desarrollado nuevos avances tecnológicos que han dado como resultado el aumento del número de parámetros que se pueden estudiar, incluso a nivel de célula única.
Tal y como mostramos en las newsletter anteriores, las nuevas técnicas de citometría de masas, citometría espectral, las plataformas de análisis ómicos en célula única y, cómo no, la citometría multiparamétrica convencional (de 8 parámetros en adelante), generan sets de datos muy grandes y con muchas dimensiones, tantas como parámetros se midan (“High Dimensional Data”). Esto hace que el análisis manual con técnicas clásicas manuales se complique.
Tradicionalmente, los datos de citometría de flujo se visualizan como un set de gráficos de 2 dimensiones, donde se dibujan una serie de regiones de interés (gates) que definen poblaciones, las cuales se examinan en nuevos plots 2D con el fin de identificar nuevas subpoblaciones, realizar medidas cualitativas (si la población está presente o ausente o cuál es su proporción relativa) o cuantitativas (intensidad de fluorescencia de un determinado marcador). Al aumentar el nº de parámetros a estudio, el nº de plots 2D que tenemos que realizar para cubrir el análisis de todos los parámetros aumenta muchísimo. Por ejemplo: en un experimento con 18 colores deberían hacerse un total de 154 gráficos 2D para poder visualizar cada combinación de marcadores presentes en la muestra.
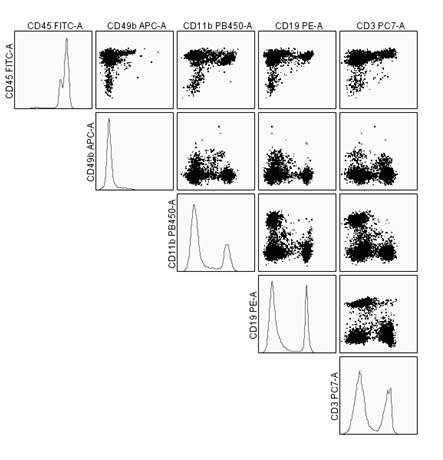
Fig.1. Para poder analizar poblaciones y subpoblaciones presentes en una muestra tenemos que analizar todos los marcadores de dos en dos. En el ejemplo, dónde se usan 5 marcadores de fluorescencia, para analizar las relaciones entre todos ellos tendríamos que realizar 10 plots. Si además lo comparamos con parámetros de dispersión, el nº de plots aumentaría a 22.
Todo esto hace que aumente la complejidad y el tiempo dedicado análisis, se corre el riesgo de no identificar subpoblaciones y de establecer un sesgo por subjetividad y variabilidad entre las estrategias de gating realizadas por distintas personas. Esto puede hacer que información potencialmente relevante no sea reconocida y por tanto se ignore.
Para intentar paliar estos problemas y avanzar en el análisis de datos, se han desarrollado nuevos algoritmos automatizados que exploran la estructura de los datos y los agrupan en función de sus características.
En esta ocasión vamos a hablar de modelos de reducción dimensional y de clustering.
REDUCCIÓN DIMENSIONAL
Las técnicas de reducción dimensional permiten visualizar los datos encontrando una representación de menores dimensiones, normalmente dos, preservando la estructura del espacio multidimensional.
Uno de los métodos más utilizado en citometría es el tSNE: t-distributed Stochastic Neighbour Embedding. El otro método de reducción dimensional más utilizado es el UMAP: Uniform Manifold Approximation and Projection.
Estos algoritmos encuentran las similitudes de células en el espacio multidimensional, es decir, encuentran células que son similares en cuanto a los todos los marcadores (dimensiones) que expresan, así como en los niveles de expresión de estos, y las muestra en un plot de 2 dimensiones denominado mapa tSNE (o UMAP). La proximidad de las células en este mapa refleja sus distancias en el espacio multidimensional, de forma que células que tienen patrones de expresión similares están localizadas muy cerca.
Fig.2: Ejemplo de tSNE maps. Cada nube o isla de puntos indica una población con patrones de expresión similares.
Lo primero que hacen es computar los datos que se encuentran más próximos entre sí en el espacio multidimensional mediante análisis por pares y a continuación, optimiza repetitivamente el posicionamiento en un espacio 2D.
Generalmente, las herramientas de reducción dimensional, aunque te permiten una visualización más sencilla del espacio multidimensional, no proporcionan una clasificación automática de las células o su pertenencia a un clúster concreto. Por eso, debemos después validar con gating manual a qué poblaciones corresponden las distintas islas.
Otra opción es utilizar un algoritmo de clustering para que, de forma no supervisada, nos muestre los distintos clústeres que encuentra en nuestra muestra.
CLUSTERING
Los algoritmos de clustering van a identificar poblaciones y subpoblaciones de células con expresión similar de marcadores, sin necesidad de intervención por parte del investigador. Aunque al igual que ocurre con el tSNE, será necesaria la validación de los clústeres identificados por el algoritmo mediante gating manual.
Estos algoritmos son objetivos, ya que no hacen asunciones de qué poblaciones estamos esperando, sino que analizan la expresión de todos los marcadores en cada una de las células de la muestra y a continuación las agrupan en clústeres según la expresión de los mismos. De esta manera van a permitir la detección de poblaciones no esperadas.
Existen diversos algoritmos: FlowMerge, FlowSOM, SPADE, Citrus, X-Shift etc. La elección de utilizar uno o de otro dependerá de cuál se ajusta mejor a los datos que nosotros conocemos en cuanto a poblaciones diferentes que podemos esperar. Es decir, el algoritmo debe encontrar las poblaciones básicas que existen en nuestra muestra.
Vamos a ver un ejemplo sencillo para mostrar estos algoritmos.
EJEMPLO
Tomamos unos datos obtenidos a partir de células de bazo de ratón C57BL/6 (sí….son células inmunes, pero esto se puede aplicar a células del sistema nervioso también), teñidas con 5 marcadores fluorescentes que nos van a definir varias poblaciones según el gating manual:
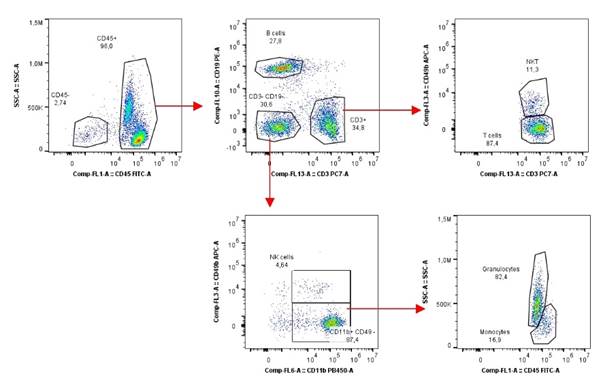
Fig. 3: Gating manual. Podemos definir 7 poblaciones únicas: Linfocitos T, linfocitos B, células NK, células NKT, granulocitos, monocitos y células CD45 neg.
Si aplicamos los algoritmos de reducción dimensional tSNE y UMAP sobre esta misma muestra obtenemos los siguientes gráficos:
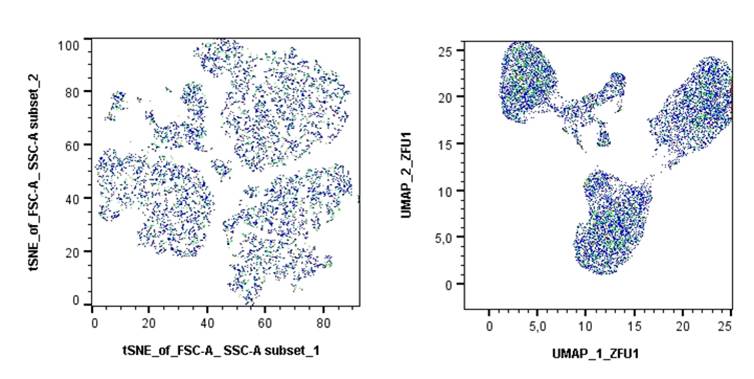
Fig.4: Cada isla nos indica agrupación de células con las mismas características
Podemos analizar la expresión de cada marcador en estas representaciones para conocer dónde se encuentran nuestras poblaciones principales:
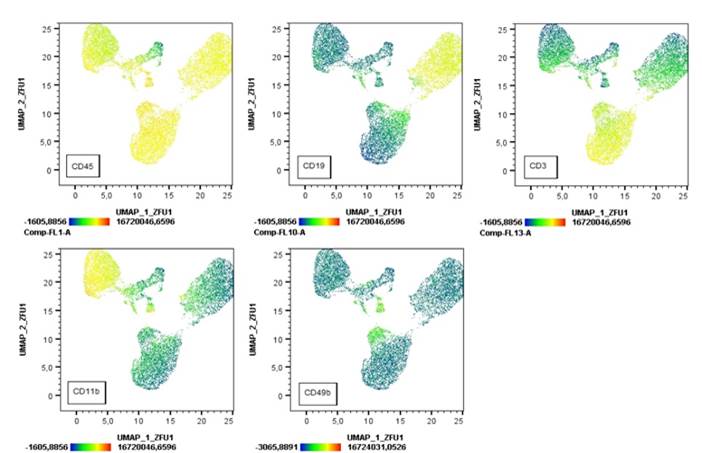
Fig. 5: Mapas de calor indicando la intensidad de fluorescencia de cada marcador (indicado en el recuadro de cada gráfico). Podemos identificar qué nube/isla expresa por ejemplo el marcador de células B CD19, o el marcador CD3 presente en células T.
Si utilizamos un algoritmo de clustering como FlowSOM sobre esta muestra, este algoritmo nos identifica hasta 8 clústeres diferentes:

Fig.6. A la izquierda, representación de los clústeres encontrados por el algoritmo FlowSOM representado sobre mapa tSNE. A la derecha, solapamiento de las poblaciones encontradas mediante gating manual, representadas sobre mapa tSNE.
Como se puede observar por los colores, el algoritmo FlowSOM es capaz de detectar las poblaciones principales sin supervisión, e incluso encontrar poblaciones que no definimos con gating manual (marcadas con un círculo rojo). Sin embargo, el algoritmo es mejorable porque no es capaz de diferenciar células NKT con respecto a la población total de células CD3+ (marcado en azul).
Si utilizamos estos algoritmos sobre muestras tratadas/no tratadas o procedentes de distintos pacientes o controles, podemos encontrar diferencias entre poblaciones que aparecen/desaparecen en las distintas condiciones. Como último ejemplo, esta comparativa entre muestras no tratadas y muestras tratadas
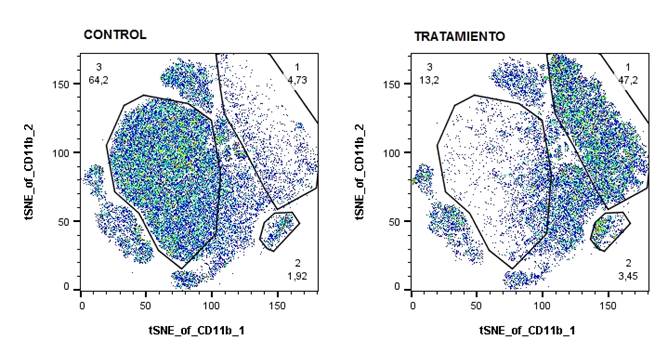
Fig.7: Representación tSNE. Se puede ver como el tratamiento induce la desaparición de la población 3 y la aparición de la población 1, entre otros cambios no mostrados. Esto indica un cambio en las características de esas poblaciones inducido por el tratamiento.
Lecturas recomendadas:
– Cossariza A et al (2019) Eur. J. Immunol: https://doi.org/10.1002/eji.201970107
– Galli E et al (2019) Eur.J. Immunol: https://doi.org/10.1002/eji.201847758