Las recientes publicaciones [1],[2] en las que se resumen fundamentos y estrategias de los conceptos básicos de proteómica basada en espectrometría de masas, nos han hecho recapitular y revisar un poco los recursos y lecturas de interés para aquellos que se inicien en este mundo de la proteómica.
Por ello hacemos una revisión de nuestras fuentes y os dejamos unos cuantos enlaces y reseñas para ir introduciéndose en la espectrometría de masas, los procesos de preparación de muestras y las estrategias de cuantificación y análisis.
Fundamentos de la espectrometría de masas
La espectrometría de masas (MS) es un método que puede analizar una variedad de compuestos químicos y biológicos. Los espectrómetros detectan la presencia y abundancia de biomoléculas (péptidos, proteínas, metabolitos y lípidos) o de productos de síntesis utilizando propiedades fundamentales de las moléculas, como la masa y la carga neta.
Todos los espectrómetros de masas tienen tres componentes fundamentales: una fuente de ionización, un analizador de masas y un detector.

Fig. 1 Componentes básicos de un espectrómetro de masas típico. * FT-ICR no utiliza un multiplicador de electrones. APCI, ionización química a presión atmosférica; ESI, ionización por electropulverización; MALDI, desorción / ionización láser asistida por matriz; FT-ICR, resonancia ion-ciclotrónica de Fouriertransform.
Una de las principales páginas para conocer los fundamentos de la espectrometría de masas es la página creada por la American Society for Mass Spectrometry (ASMS) en ella encontrareis grandes charlas.
Podéis encontrar links interesantes en YouTube donde se muestra cada una de las partes de los espectrómetros y los diferentes tipos de modos de ionización (ESI, MALDI, APCI, EI…), tipos de analizadores (tiempo de vuelo, cuadrupolo, trampa de iones, orbitrap) o una combinación de ellos (analizadores en tándem), y detectores. Las casas comerciales suelen publicar en sus canales videos mostrando sus últimos avances. Os recomendamos los canales del Broad Institute, IIT Bombay entre otros.
Uno de nuestros recursos educativos favoritos es la página de IonSource, en ella podéis encontrar diversos tutoriales sobre identificación, cuantificación y una gran recopilación de enlaces para definiciones, referencias, historia, Instituciones, etc.
Otro de los enlaces interesantes es al glosario de términos del Kings College de Londres, ya que la terminología utilizada está llena de siglas y acrónimos que debemos traducir para aquellos que se inician en este campo.
Para reseñas más antiguas, pero con descripciones muy completas tenéis la revisión de Glish, G., et al[3], Graves, PR., et al[4] y de la NIAAA donde se comienza desde la perspectiva genómica / transcriptómica y va avanzando.
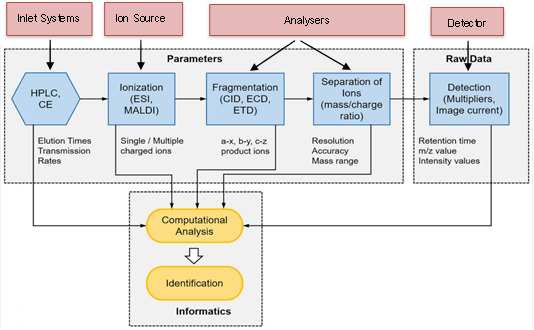
Fig. 2 Resumen de la información básica generada durante los experimentos basados en MS. Las flechas muestran la información experimental recopilada de cada paso del MS. Esta información es crucial para permitir una identificación precisa de proteínas mediante análisis computacional.
Si queréis profundizar aún más, os engancha la interpretación de espectros y las reglas de fragmentación, necesitareis como biblia “Interpretation of mass spectra” de F.W. McLafferty o una revisión un poco más abreviada por Nicolescu T.O.
AUTOR
Gemma Barroso-García, MSc.
Responsable del Servicio de Proteómica.
Hospital Nacional de Parapléjicos.
Toledo, España.
REFERENCIAS
[1] Sinha A., Mann M. A beginner’s guide to mass spectrometry-based proteomics. Biochem (Lond). 2020; BIO20200057. Published 2020 Sep 09. https://doi.org/10.1042/BIO20200057
[2] Zainab Noor, Seong Beom Ahn, Mark S Baker, Shoba Ranganathan, Abidali Mohamedali, Mass spectrometry–based protein identification in proteomics—a review, Briefings in Bioinformatics, bbz163, https://doi.org/10.1093/bib/bbz163.
[3] Glish, G., Vachet, R. The basics of mass spectrometry in the twenty-first century. Nat Rev Drug Discov 2, 140–150 (2003). https://doi.org/10.1038/nrd1011
[4] Graves P.R., Haystead T.A.J. Molecular Biologist’s Guide to Proteomics. Microbiol. Mol. Biol. Rev. 2002;66:39–63. doi: 10.1128/MMBR.66.1.39-63.2002